- 2012-06-28 (木) 19:30
- DBCLS
6/12-20の期間、東京大学理学部生物化学科・生物情報科学科3年生の学生実習を担当しました。テーマは「マイクロアレイを用いた網羅的遺伝子発現解析」。生物情報では、2009年にAgilent社のマイクロアレイ設備一式を学生実習のために導入しており、学科の共通機器として毎年の学生実習に使用しているほか、各ラボの研究にも活用されています。調達時にAffymetrixとAgilentの方に伺ったところ、学部の学生実習でマイクロアレイをやるところは聞いたことがないとのこと。実習のプロトコルも試行錯誤を重ね、今年で4年目になります。
miRNAによる標的遺伝子の抑制
実習では、ヒトのHeLa細胞にマイクロRNA (miRNA) を導入して24時間後に回収したものを各班に配布。miRNA導入による遺伝子発現の変動を、マイクロアレイで調べるのがねらいです。
miRNAは、5′末端から2-8の位置の ‘seed’ と呼ばれる領域をたよりにmRNAの3′ UTRと結合し、翻訳抑制やmRNAの分解を誘導することが知られています。
ヒトのlet-7b:5′-UGAGGUAGUAGGUUGUGUGGUU-3′ (miRBaseより。赤字部分がseed)
実習で使用したmiRNAとは異なりますが、たとえばヒトのmiRNAのひとつであるlet-7bの場合は、赤字で示した GAGGUAG がseedと呼ばれる領域です。これと相補的な CUACCUC という配列を3′ UTRにもつ (以下、seedマッチする と表現) mRNAは、let-7bの標的となりその発現が抑制される可能性があります。ただし、seedマッチするmRNAのすべてが標的となるわけではなく、またseedマッチ以外にもさまざまなfactorが標的の認識および抑制に寄与することがわかっています。miRNAの標的予測については、TargetScan、PicTar、miRandaなどさまざまなプログラムが開発されていますが、いずれもfalse positiveが多く、正確な予測は難しいのが現状です。
seedマッチする遺伝子の変動
実習では1日目に細胞からtotal RNAを抽出、2日目に逆転写とcRNA合成、3日目にアレイへのハイブリダイゼーション、4日目にアレイのスキャンをおこない、4〜6日目の3日間でデータの解析をしました。この3日間は情報基盤センターの演習室をお借りしました。
データの解析には、以下のソフトを利用しました。
- Excel – マイクロアレイのデータ解析をブラックボックスにしないために、まずは生の数値にきちんと触れてもらいました。アレイ間の正規化をおこない各種プロットを作成。
- GeneSpring GX – エクセルで全部やるのは苦行なので、一連のデータ解析は基本的にGeneSpring GXを使用。Agilent社より学生実習のための期間限定ライセンスを供与いただきました。
- R – 今年はスムーズに進んだのでRによる解析も紹介。マイクロアレイデータ解析用のパッケージlimmaを利用し、コードの基本部分は講師が提供。
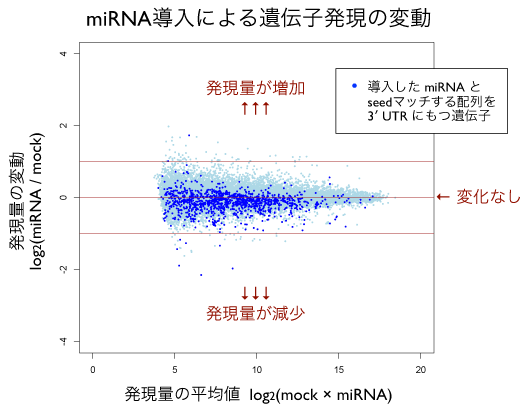
細胞に導入したmiRNAとseedマッチする遺伝子をリストアップして、それらの変動のようすをMAプロット上に可視化してみます。すると、seedマッチする遺伝子群 (青色) は、その他の遺伝子群 (水色) と比較して下側に分布しており、発現量が減少している傾向が確認できました。ただし減少というのはあくまで集団としての傾向で、個々の遺伝子に注目すると、seedマッチしていても変化がないか、増加しているような遺伝子もたくさんあります。
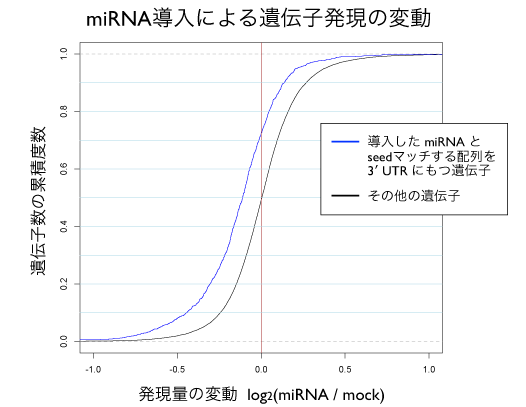
発現量が減少している傾向を、より定量的に評価してみます。seedマッチする遺伝子群 (上のMAプロットで青色)、その他の遺伝子群 (水色) のそれぞれについて、MAプロットの縦軸の値「発現量の変動」でソートしたときの累積度数を求め、プロットします。
その他の遺伝子群 (黒色の線) は、(0, 0.5) を通る対称的な曲線になりました。一方、seedマッチする遺伝子群 (青色の線) は、それよりも左方向にシフトしており、発現量が減少している傾向がはっきりわかります。もしまったく減少していなければ、黒い曲線と重なるはずです。このように、MAプロットでは減少がわかりにくい場合でも、累積度数曲線を作成すると評価しやすくなります。
miRNAのseedが修飾されると標的は変わるか?
今回の実習に使用したmiRNAは、seed領域のAがI (イノシン) に修飾されることが知られています。このときmiRNAの標的となる遺伝子群はどのように変化するのでしょうか。実習では、AをGに置換したmiRNA (G-type)、および、AをIに置換したmiRNA (I-type) についてもマイクロアレイをおこないました。
G-type を導入した細胞では、もともとのseedがマッチしていた遺伝子群は抑制されず、A → G に置換したseedがマッチする遺伝子群が抑制されるようになりました。seedが1塩基かわるだけで、標的となる遺伝子群が大きく変わってしまうことを実感できたのではないかと思います。
一方、I-type を導入した細胞では、やはりもともとのseedがマッチしていた遺伝子群は抑制されず、A → G 置換したseedがマッチする遺伝子群が少し抑制されていました。また、A → U 置換したseedがマッチする遺伝子群、A → C 置換したseedがマッチする遺伝子群は、どちらも抑制されていませんでした。
イノシンは複数種類の塩基 (C, A, U) と塩基対を形成できますが、実習の結果を考慮すると、seed領域の A → I 修飾は、A → G 置換に近い効果がありそうです。もちろんこれを一般的に示すには、今回の実習で使用した特定のmiRNAのアレイデータだけでは不十分ですし、レポーター実験などによる詳細な検討も必要でしょう。
もっとも、6日間の学生実習でここまで解析ができて発展性もありそうな結果が出たのはよかったのではないかと思います。
- Newer: DDBJ 89.0 (Jun, 2012)
- Older: DDBJの塩基配列データベース
Trackbacks:0
- Trackback URL for this entry
- /archives/1876/trackback
- Listed below are links to weblogs that reference
- マイクロアレイ学生実習 from mesoの実験ノート