Home > Archives > 2011-05
2011-05
生物種の追加とデータベース更新
- 2011-05-31 (火)
- DBCLS
前回のpostでは、mesoが構築している「遺伝子をGoogleのように(?)検索できるサイト」を紹介した。
http://bit.ly/RNAbrowser→ http://GGRNA.dbcls.jp/ (8/18更新:URLを変更)
まずはヒト遺伝子バージョンを作って公開したわけだが、それだけではヒトをやっている人にしか使ってもらえないので生物種を増やすことにした。とは言ってもマシンの容量の制約があるので、取り入れる生物種を絞らないといけない。
生物種の選定
今回はmesoの独断と偏見に基づき、モデル生物として普及しており、かつゲノムや個々の遺伝子に関する情報が豊富な生物種を10種くらい選ぶことにした。@synobuさんが教えてくれたGene Ontologyコンソーシアムのページも参考にした。
- GO: Reference Genome Annotation Project – http://www.geneontology.org/GO.refgenome.shtml
このページの中ほどにあるモデル生物のリストが使えそう。
- Arabidopsis thaliana – シロイヌナズナ
- Caenorhabditis elegans – 線虫
- Danio rerio – ゼブラフィッシュ
- Dictyostelium discoideum – タマホコリカビ(今回は見送り)
- Drosophila melanogaster – ショウジョウバエ
- Escherichia coli – 大腸菌(今回は見送り)
- Gallus gallus – ニワトリ
- Homo sapiens – ヒト
- Mus musculus – マウス
- Rattus norvegicus – ラット
- Saccharomyces cerevisiae – 出芽酵母
- Schizosaccharomyces pombe – 分裂酵母
このうち大腸菌、タマホコリカビ、それから別にリクエストをいただいた枯草菌、シアノバクテリアは、データの形式の問題で今回は見送り、別に対応を考えることにした。またtwitterでご連絡いただいたイネをはじめNBRPな生物種は次回以降の更新でぜひ取り入れたい。
データの取得
ちょうど5月に入ってRefSeq release 47がリリースされたので、ついでにヒトの配列も更新する。RefSeqは2ヶ月に1回の頻度で新しいバージョンがリリースされるので、mesoの検索サイトもそれにあわせて更新できるよう、データベースの追加・更新を簡単にできるような仕組みをつくる必要がある。
RefSeqの最新版は下記からダウンロードできる。
この中にある全生物種の転写産物を収録したGBFF(GenBank Flat File)形式のファイル、completeXXX.rna.gbff.gz(release 47ではXXXは1〜418)をダウンロードした。容量はgz圧縮で3.4GB、展開すると17.2GBある。各transcriptのSOURCE欄を見て特定の生物種だけを抜き出すスクリプト(grep_gbff.pl)を使い、前述の10種を抜き出した。
| 生物種 | GenBank形式(GBFF) ファイルのSOURCE欄 | Transcript数 | GenBank形式(GBFF) ファイルの容量(MB) |
|---|---|---|---|
| ヒト | Homo sapiens (human) | 46,727 | 846.6 |
| マウス | Mus musculus (house mouse) | 35,892 | 585.4 |
| ラット | Rattus norvegicus (Norway rat) | 30,862 | 225.0 |
| ニワトリ | Gallus gallus (chicken) | 5,418 | 38.8 |
| ゼブラフィッシュ | Danio rerio (zebrafish) | 28,261 | 190.5 |
| ショウジョウバエ | Drosophila melanogaster (fruit fly) | 22,929 | 352.1 |
| 線虫 | Caenorhabditis elegans | 24,377 | 165.0 |
| シロイヌナズナ | Arabidopsis thaliana (thale cress) | 34,525 | 197.0 |
| 出芽酵母 | Saccharomyces cerevisiae S288c | 5,863 | 37.8 |
| 分裂酵母 | Schizosaccharomyces pombe 972h- | 5,010 | 39.6 |
| 合計 | 239,864 | 2677.7 |
検索サイト更新
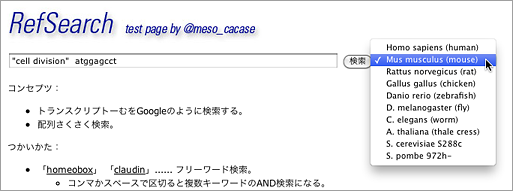
以上のデータを取り入れて検索サイトをアップデートした。当初はあくまで「窓ひとつ」で検索できることにこだわっており、spe:human のように生物種もタグで指定することを考えていたが、それはやめてプルダウンで選ぶ方針にした。ユーザの立場で考えてみると、検索の時点で生物種が決まっている場合が多いだろうということと、普段ショウジョウバエなど特定のモデル生物を扱っている研究者は、その種を中心に検索することが多いという事情を考慮した。
どうせなら入口を分けてもよいかもしれない。下記のURLからアクセスすると、最初からそれぞれの種がプルダウンで選ばれた状態になる。毎回プルダウンを選択する必要がないうえに、ブックマークもしやすいだろう。
- ヒト → http://GGRNA.dbcls.jp/hs/
hsを省略して http://GGRNA.dbcls.jp/ だけでもOK - マウス → http://GGRNA.dbcls.jp/mm/
- ラット → http://GGRNA.dbcls.jp/rn/
- ニワトリ → http://GGRNA.dbcls.jp/gg/
- ゼブラ → http://GGRNA.dbcls.jp/dr/
- ハエ → http://GGRNA.dbcls.jp/dm/
- 線虫 → http://GGRNA.dbcls.jp/ce/
- シロイヌナズナ → http://GGRNA.dbcls.jp/at/
- 出芽酵母 → http://GGRNA.dbcls.jp/sc/
- 分裂酵母 → http://GGRNA.dbcls.jp/sp/
(6/1更新)URLを少し変更して /~meso/ なしでアクセスできるようにしました。過去にアナウンスしたURLも当分は使えますが、上記のURLをご利用ください。
(8/18更新:URLを全面的に変更しました。過去にアナウンスしたURLから転送されます。
ちなみにこれはウェブサーバ側でURLの /mm/ の部分を index.cgi?spe=mm などと書き換えるRewriteRuleを書いて実現している。mod_rewrite便利。
さて、こうしているうちに新たなバグが見つかったので修正しないと。
- Comments (Close): 0
- Trackbacks: 0
遺伝子をGoogleのように検索できるサイト
- 2011-05-18 (水)
- DBCLS
遺伝子をGoogleのような感覚でサクサク検索できたら便利だろうと考え、サイトをつくってみた。mesoが構築している「RNA統合データベース」を構成する仕組みのひとつとなる予定だ。
http://bit.ly/RNAbrowser→ http://GGRNA.dbcls.jp/ (8/18更新:URLを変更)
NCBI RefSeq release 46 (Mar, 2011)に登録されているヒトのtranscriptを対象にフリーワードで検索できる。とくに、塩基配列まで含めて検索できるようにしたところがこのサイトの売りで、オプションにより1〜2塩基のミスマッチを許した検索も可能だ。
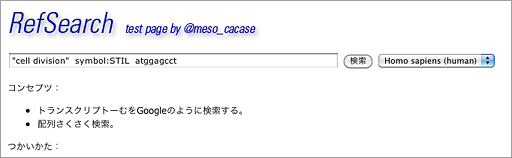
使いかたは、検索ウィンドウに単語を打ち込むだけ。あえて入り口はシンプルにした。細かい条件をつけたいときのために、aa:KLQEEM(アミノ酸配列から検索)のようなタグを何種類か用意した。ただしタグを明示的につけなくても多くの場合は望みの結果が得られるはずだ。
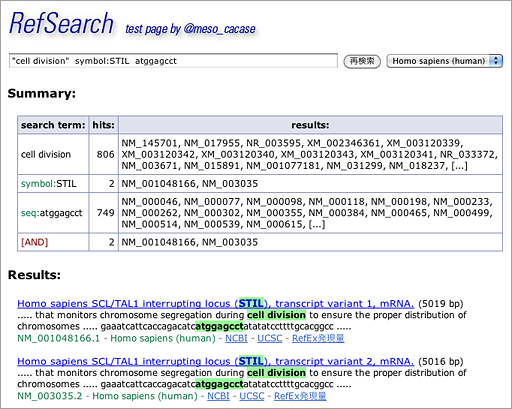
複数のキーワードを入れるとAND検索になり、たとえばこんな結果が表示される。
上の例では、”cell division”というキーワードで806件、symbol:STIL(symbolから検索)で2件、atggagcctという塩基配列を持つものが749件。そして、3つの検索語をすべて含むものが2件([AND]欄を参照)見つかった。Results:欄にその2件の遺伝子の概要が表示されている。
(5/26追記)「2件の遺伝子」と書いてしまったが、この2件はSTILという1つの遺伝子に由来する2種類のtranscript variantである。
このように複数のキーワードを入れた場合は、いわゆる絞り込み検索をするのではなく、3つの独立した検索をおこなってその結果の共通部分を求める仕組みにした。これは、各々の検索語ごとのヒット件数がわかるほうが便利だろうと思ったからだ。たとえば、ユーザの意図する検索結果が出てこなかった場合、検索語を変えたうえで再検索することになるわけだが、どの検索語を変えればうまく絞り込めるのかを考える上で参考になるだろう。
キーワードごとに独立した検索をおこなっているため時間はかかるが、それでも上記の結果は約1秒で返ってくる。
さて、Results:欄だが、NCBIのnucleotide、UCSCゲノムブラウザ、RefExにリンクしている。RefExはDBCLSで提供している発現量のリファレンスデータセットだ。検索でヒットした遺伝子がどのくらい発現しているのかをワンクリックで概観できる。
- RefEx — http://togoexp.dbcls.jp/aboutrefex
DBCLSが提供している遺伝子の発現量データベース。
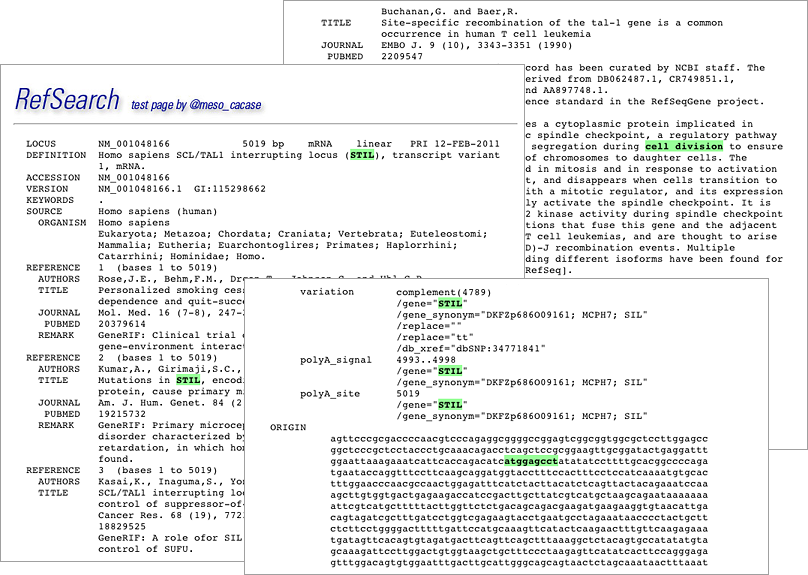
さらに、Results:欄の各遺伝子のリンクをクリックすると、おなじみのGenBankフォーマットで詳細が表示される。入力したキーワードがハイライトされ、見つけやすくなっている。
開発途上のため不安定なところがあるかもしれない。お手数でなければ不具合のご指摘やコメントなど頂ければたいへんありがたい。
将来は、転写産物=RNAをベースに知識(塩基配列、文献、プローブ、siRNA、等々)を統合したデータベースをつくり、さまざまな切り口で遺伝子を「さがす」または「ながめる」ことができるようなサイトの公開を目指している。何をどんなふうに統合して、どんな使い勝手になるのかは別に紹介していきたいと思う。今回はその準備という位置づけだ。
なお今回のサイトは、2006年頃に私がPreferred Infrastructureの西川徹さんらと一緒にコンセプトや仕様を考え、西川さんらが実装したGenome Sedueというサイトをもとにして、RNA統合データベースに必要な仕組みを盛り込んで発展させている。
- Preferred Infrastructure — http://preferred.jp/
検索エンジン関連を中心に非常に高い技術をもつ会社。 - Genome Sedue — http://labs.preferred.jp/genome/
現在は使えなくなっている。
- Comments (Close): 0
- Trackbacks: 0
Home > Archives > 2011-05
- Search
- Feeds
- Meta