Home > Archives > 2011-06
2011-06
統合遺伝子検索GGRNA
- 2011-06-27 (月)
- DBCLS
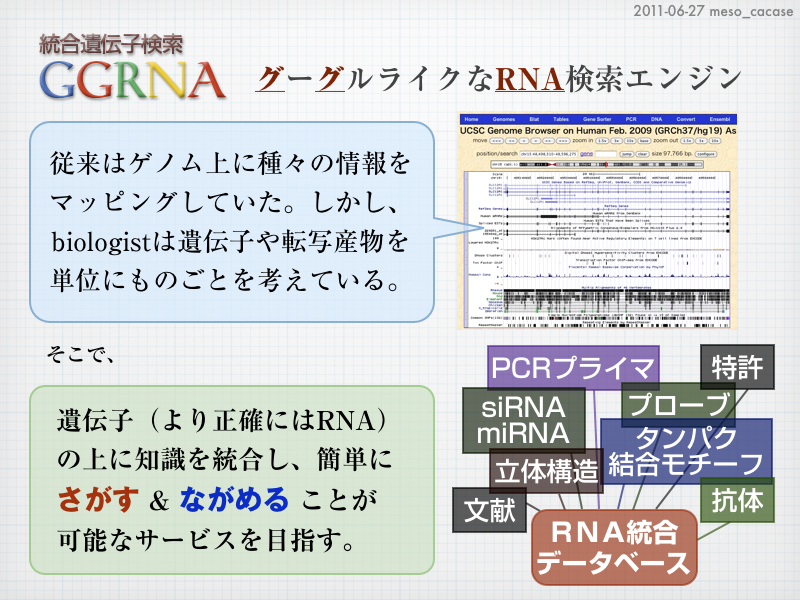
mesoが構築している「遺伝子をGoogleのように(?)検索できるサイト」の名前を「GGRNA」にしてみました。表向きはグーグルライクなRNA検索エンジンでGGRNA。しかし裏では “ググるな” と呼んでいたりします。
これに伴いURLも変更しました。今までのURLも生きていますが、今後はこちらをお使いください。
- 統合遺伝子検索GGRNA —
http://bit.ly/GGRNA→ http://GGRNA.dbcls.jp/ (8/18更新:URLを変更)
下記は定例会議で報告した資料の一部。研究総括からは「Googleに怒られないか?」というご指摘が(爆 もし怒られたら変えます。。。
資料の2枚目は今後の方針について。GGRNAは遺伝子を「さがす」仕組みを実装したもの。それと同じくらい大切なのが、ヒットした遺伝子を「ながめる」仕組み。短い配列や立体構造、文献などのさまざまな知識をRNAのうえに整理して見せたいのですが、その見せ方の部分を工夫したいと思っており、ある程度形になったらここでご紹介します。
- Comments (Close): 0
- Trackbacks: 0
siDirectで大量のsiRNAを設計する
- 2011-06-20 (月)
- DBCLS
mesoが東大生化で開発していたsiRNA設計サーバ・siDirectを使って大量にsiRNAを設計したいというリクエストをいただいた。siDirectは、RNAi活性が高く(つまり良く効く)、標的以外の遺伝子が意図せず抑制される「オフターゲット効果」が少ないsiRNAを効率よく設計できるウェブサーバである。
- siDirect 2.0 — http://siDirect2.RNAi.jp/
- @_junk_0 さん作の siDirect 2.0 解説動画
実はそういう要望は以前からときどき頂いていたので、この機会にsiDirectのウェブをcrawlするスクリプトを書いてみた。
- siDirect2crawl.pl (gist.github)
つかいかた:
% ./siDirect2crawl.pl sequence.txt
sequence.txtは、FASTAファイルもしくは塩基配列だけを格納したテキストファイル。ATGCU以外の文字は無視(削除)される。大文字小文字は区別しないが、結果のtarget sequenceの欄は入力ファイルの大文字小文字が反映される。WWW::Mechanizeというモジュールを使用しているので、あらかじめCPANからインストールする必要がある。
結果の例:
[siDirect v.2.0 | 2011-06-20 18:07:26]
target position target sequence RNA oligo, guide passenger functional siRNA selection seed-duplex stabilty (Tm), guide passenger min. number of mismatches against off-targets, guide passenger
24-46 cagaagaatggtacaaatccaag UGGAUUUGUACCAUUCUUCUG GAAGAAUGGUACAAAUCCAAG U 20.1 12.0 2 2
159-181 cccttaaaggaaccaatgagtcc ACUCAUUGGUUCCUUUAAGGG CUUAAAGGAACCAAUGAGUCC U 18.1 11.0 2 2
261-283 aggatgagattcagaatatgaag UCAUAUUCUGAAUCUCAUCCU GAUGAGAUUCAGAAUAUGAAG U 8.7 20.4 2 3
461-483 ctggttgatacccactcaaaaag UUUUGAGUGGGUAUCAACCAG GGUUGAUACCCACUCAAAAAG U 19.2 16.1 3 2
462-484 tggttgatacccactcaaaaagg UUUUUGAGUGGGUAUCAACCA GUUGAUACCCACUCAAAAAGG U 12.2 16.1 3 2
491-513 ctgattaagacggttgaaactag AGUUUCAACCGUCUUAAUCAG GAUUAAGACGGUUGAAACUAG U 14.9 6.9 3 3
519-541 gacaggttatcaacgaaacttct AAGUUUCGUUGAUAACCUGUC CAGGUUAUCAACGAAACUUCU U 19.7 18.5 3 3
521-543 caggttatcaacgaaacttctca AGAAGUUUCGUUGAUAACCUG GGUUAUCAACGAAACUUCUCA U 13.3 16.1 2 3
このようにsiDirectのweb版でテーブルとして表示される部分が、タブ区切りテキストで得られる。
siRNAを設計する際のオプションは、スクリプト内の %param で与えることができる。詳細は下の図およびコードを参照。
大量にクエリを投げる場合の例。サーバに負荷が掛からないよう、sleepコマンドをはさんでほしい。
% ls
input_sequences/ siDirect2crawl.pl* siDirect_result/
% cd input_sequences/
% ls
NM_000014.fa NM_000015.fa NM_000016.fa […]
% foreach n ( * )
foreach> ../siDirect2crawl.pl $n > ../siDirect_result/$n.siRNA
foreach> sleep 5
foreach> end
[…]
% cd ../siDirect_result/
% ls
NM_000014.fa.siRNA NM_000015.fa.siRNA NM_000016.fa.siRNA […]
- Comments (Close): 0
- Trackbacks: 0
RefExとの連携&データ出力機能を追加
- 2011-06-13 (月)
- DBCLS
RefExとの連携
マイクロアレイのプローブIDから配列をつかって遺伝子を検索できるようにしたので、RefExからリンクを張っていただきました。RefExはDBCLSで提供している発現量のリファレンスデータセット。これで相互リンクが実現。同じ部屋の方の仕事とリンクできていい感じです。
データ出力機能
検索結果を外部のソフトで利用しやすいように、タブ区切りテキストで出力できるようにしました。
「タブ区切りテキスト。エクセルとかに直接コピペできます。エクセルはお嫌いですか?→ Numbersへ」
窓の中身を選択して、たとえばExcelに直接コピー&ペーストすれば
うまい具合にセルに入ります。 もちろんNumbersでもうまくいきますよ!!
- Comments (Close): 0
- Trackbacks: 0
アレイのプローブ配列検索を強化
- 2011-06-09 (木)
- DBCLS
マイクロアレイのプローブIDから塩基配列検索の機能を強化しました。
「便利そう」なサービスを実際に「使える」サービスにするためには、地味な作り込みが不可欠なんですよね・・・。先週紹介したプローブ配列検索も、ヒトとマウスで合計4つのプラットフォームだけでは頼りない。そこで今回は、AffymetrixとAgilentのマイクロアレイのうち、GEOにサンプル登録が多いプラットフォームと、まだサンプル登録は少ないけれども最新のプラットフォームを、とにかく全部収録することに。
収録したプラットフォーム一覧
| メーカー | 生物種 | マイクロアレイの種類 | GPL ID |
|---|---|---|---|
| Affymetrix | ヒト | Human Genome U219 Array | GPL13667 |
| Affymetrix | ヒト | Human Genome U133 Plus 2.0 Array | GPL570 |
| Affymetrix | マウス | Mouse Genome 430 2.0 Array | GPL1261 |
| Affymetrix | ラット | Rat Genome 230 2.0 Array | GPL1355 |
| Affymetrix | ラット | Rat Genome U34 Set (U34A/B/C) | GPL85,GPL86,GPL87 |
| Affymetrix | ニワトリ | Chicken Genome Array | GPL3213 |
| Affymetrix | ゼブラフィッシュ | Zebrafish Genome Array | GPL1319 |
| Affymetrix | ショウジョウバエ | Drosophila Genome 2.0 Array | GPL1322 |
| Affymetrix | ショウジョウバエ | Drosophila Genome Array | GPL72 |
| Affymetrix | 線虫 | C. elegans Genome Arra | GPL200 |
| Affymetrix | シロイヌナズナ | Arabidopsis ATH1 Genome Array | GPL198 |
| Affymetrix | 出芽酵母+分裂酵母 | Yeast Genome 2.0 Array | GPL2529 |
| Affymetrix | 出芽酵母 | Yeast Genome S98 Array | GPL90 |
| Agilent | ヒト | SurePrint G3 Human Exon 2x400K Microarray (028680) | |
| Agilent | ヒト | SurePrint G3 Human Exon 4x180K Microarray (028679) | |
| Agilent | ヒト | SurePrint G3 Human GE 8x60K Microarray (028004) | GPL13607 |
| Agilent | ヒト | Whole Human Genome Microarray 4x44K v2 (026652) | GPL10332,GPL13497 |
| Agilent | ヒト | Whole Human Genome Microarray 4x44K (014850) | GPL4133,GPL6480,GPL9822 |
| Agilent | マウス | SurePrint G3 Mouse Exon 2x400K Microarray (028727) | |
| Agilent | マウス | SurePrint G3 Mouse Exon 4x180K Microarray (030493) | |
| Agilent | マウス | SurePrint G3 Mouse GE 8x60K Microarray (028005) | GPL10787 |
| Agilent | マウス | Whole Mouse Genome Microarray 4x44K v2 (026655) | GPL10333,GPL11202 |
| Agilent | マウス | Whole Mouse Genome Microarray 4x44K (014868) | GPL4134,GPL7202 |
| Agilent | ラット | SurePrint G3 Rat Exon 2x400K (028728) | |
| Agilent | ラット | SurePrint G3 Rat Exon 4x180K (028744) | |
| Agilent | ラット | SurePrint G3 Rat GE 8x60K Microarray (028279) | |
| Agilent | ラット | Whole Rat Genome Microarray 4x44K v3 (028282) | |
| Agilent | ラット | Whole Rat Genome Microarray 4x44K (014879) | GPL4135,GPL7294 |
| Agilent | ニワトリ | G. gallus (Chicken) Oligo Microarray v2 (026441) | |
| Agilent | ニワトリ | Chicken Gene Expression Microarray (015068) | GPL8764 |
| Agilent | ゼブラフィッシュ | D. rerio (Zebrafish) Oligo Microarray V3 (026437) | |
| Agilent | ゼブラフィッシュ | D. rerio (Zebrafish) Oligo Microarray (013223) | GPL2878,GPL7244 |
| Agilent | ゼブラフィッシュ | Zebrafish (v2) Gene Expression Microarray (019161) | GPL6457,GPL7301 |
| Agilent | ゼブラフィッシュ | Zebrafish Gene Expression Microarray (015064) | GPL6563,GPL7302 |
| Agilent | ショウジョウバエ | D. melanogaster (FruitFly) Oligo Microarray - V2 (021791) | |
| Agilent | ショウジョウバエ | Drosophila Gene Expression Microarray (018972) | GPL6385,GPL7300 |
| Agilent | 線虫 | C. elegans Oligo Microarray (012795) | GPL2875,GPL7272 |
| Agilent | 線虫 | C. elegans (V2) Gene Expression Microarray (020186) | GPL10094,GPL11346 |
| Agilent | 線虫 | C. elegans Gene Expression Microarray (015061) | GPL7727,GPL8209 |
| Agilent | シロイヌナズナ | Arabidopsis 3 Oligo Microarray (012600) | GPL2871,GPL7270 |
| Agilent | シロイヌナズナ | Arabidopsis 2 Oligo Microarray (V2) (013324) | GPL2880,GPL7290 |
| Agilent | シロイヌナズナ | Arabidopsis 2 Oligo Microarray (011839) | GPL888,GPL7265 |
| Agilent | シロイヌナズナ | Arabidopsis (V4) Gene Expression Microarray (021169) | GPL9020,GPL12621 |
| Agilent | シロイヌナズナ | Arabidopsis (V3) Gene Expression Microarray (015059) | GPL6177,GPL7299 |
| Agilent | 出芽酵母 | Yeast microarray (011447) | GPL884,GPL7259 |
| Agilent | 出芽酵母 | Yeast Oligo Microarray (V2) (013384) | GPL2883,GPL7293 |
| Agilent | 出芽酵母 | Yeast (V2) Gene Expression Microarray (016322) | GPL9825,GPL10045,GPL11488,GPL13340 |
| Agilent | 出芽酵母 | Yeast (V1) Gene Expression Microarray (015072) | GPL7542,GPL9294 |
プローブ検索、既知の問題点
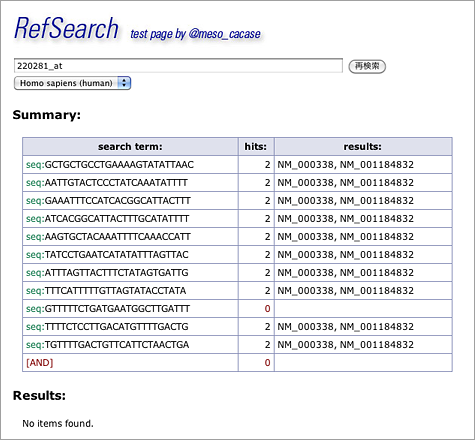
プローブを検索しても結果が何も出てこない場合があります。たとえばヒトの 220281_at というプローブ。
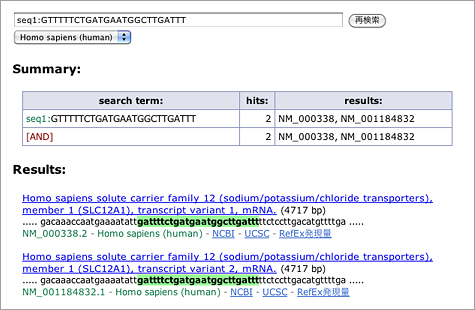
Summary欄の表をみると、11個あるプローブのうち10個は NM_000338, NM_001184832 にマッチしているものの、GTTTTTCTGATGAATGGCTTGATTT というプローブは何もヒットしていません。したがってANDを取ると何も出てこないことになります。ところがこのプローブ、1ミスマッチを許して seq1:GTTTTTCTGATGAATGGCTTGATTT で検索してやると(リンク)、ちゃんと NM_000338, NM_001184832 がヒットするのです。Affymetrix社がプローブ設計時に使った配列が、RefSeqの最新の配列と1塩基違っていたのですね。
こういう事例を想定して、本当は11個の塩基配列を検索するときに最初からミスマッチを許して seq1: とか seq2: のモードで検索すればよいのでしょうが、計算時間がかかるのでどうすべきか・・・。
- Comments (Close): 0
- Trackbacks: 0
マイクロアレイのプローブIDから塩基配列検索
- 2011-06-02 (木)
- DBCLS
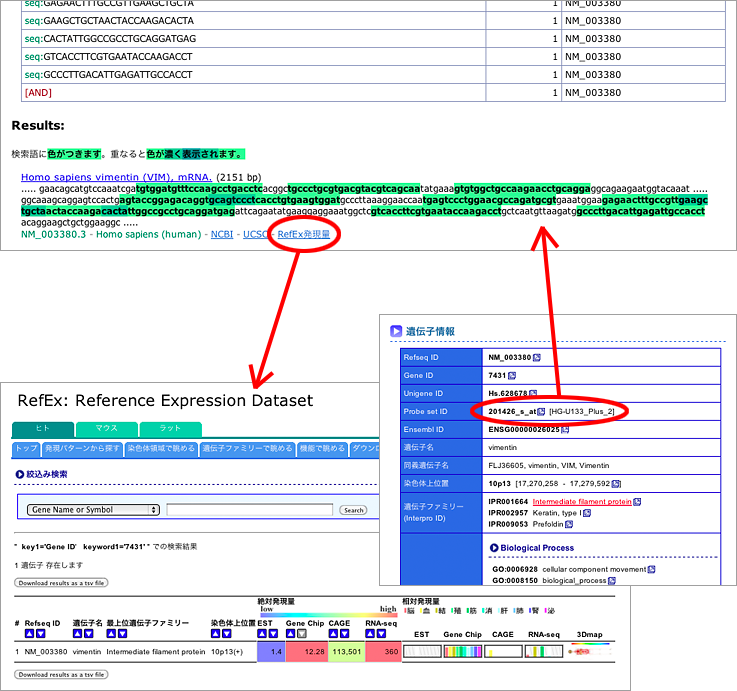
地味ですが新しい機能を追加しました。マイクロアレイのプローブIDを入力すると、そのプローブの塩基配列をつかって遺伝子を検索してくれる機能です。
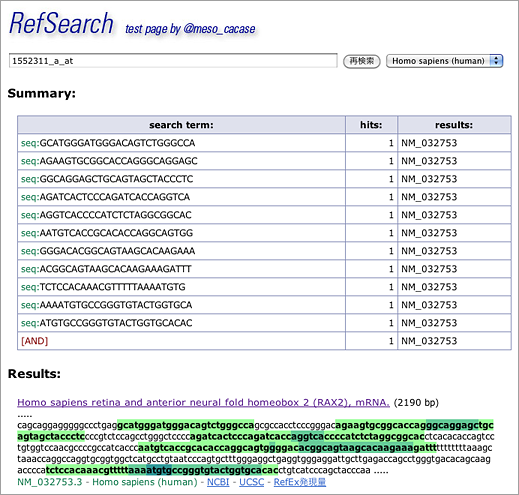
たとえば下の例は、Affymetrix社のヒトのアレイ(GeneChip Human Genome U133 Plus 2.0 Array)のプローブである、1552311_a_at で検索したものです(リンク)。Affymetrixのアレイは25-merのオリゴ11本で1つのtranscriptを認識するようになっています。この11本をまとてprobesetと呼び、probesetごとに上記のようなIDが振られています。そこで、
「1552311_a_at」
と入力すると、サーバ側でプローブID→塩基配列に展開し、
「GCATGGGATGGGACAGTCTGGGCCA」+
「AGAAGTGCGGCACCAGGGCAGGAGC」+
「GGCAGGAGCTGCAGTAGCTACCCTC」+
「AGATCACTCCCAGATCACCAGGTCA」+
「AGGTCACCCCATCTCTAGGCGGCAC」+
「AATGTCACCGCACACCAGGCAGTGG」+
「GGGACACGGCAGTAAGCACAAGAAA」+
「ACGGCAGTAAGCACAAGAAAGATTT」+
「TCTCCACAAACGTTTTTAAAATGTG」+
「AAAATGTGCCGGGTGTACTGGTGCA」+
「ATGTGCCGGGTGTACTGGTGCACAC」
で検索を実行するようにしてみました。結果はこちら。
Result欄を見ると、RAX2 (NM_032753) という遺伝子がヒットしていることがわかります。遺伝子のリンクをクリックすると、
のように、3’末端付近に11本のオリゴの標的サイトがあることがわかります。余談ですが、検索でヒットした文字列が重なった場合は緑色が濃くなるように改良しました。今までのように単色だと、どこがどのように重なっているのか全然わからなかったので。
ところで皆さんはマイクロアレイのデータを解析するときに、プローブIDと遺伝子をどのように対応させているでしょうか。おそらく多くの方は、メーカーが出している対応表や、アレイ解析ソフトの結果をそのまま利用されていると思います。しかしこれは意外と信用できないとmesoは考えています。
少し前の話なのですが、Affymetrixのアノテーションでは 226437_at → VIM とあるのに、データがおかしいのでよく調べてみたら、実際にはまったく別の遺伝子であるYIF1Bに当たっていた、みたいな事例が頻繁にありました。
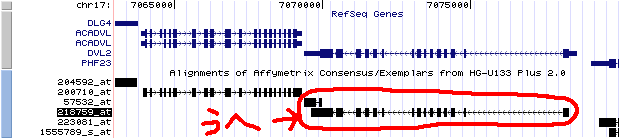
Affymetrixの表がちょっと軽くやばい感じだったので、Biomartを利用して対応表をつくることを試みたのですが、それもいまいち。例えば対応表のうえではACADVLに当たっているはずの3つのプローブが、よく調べてみると下の図のように3つのうち2つが隣の遺伝子(DVL2)に当たっていたり・・・。
上記の2つの事例に関しては、2011年6月現在は訂正されているものの、まだまだあやしい箇所が残っているのではないかと思います。だからこそ、ユーザ自身がプローブIDから塩基配列をつかって遺伝子(より正確にはtranscript)との対応を調べることは大事だと思うのです。現状ではそれを簡単にできる方法がなかったので、つくってみたというわけでした。今回は、
- GeneChip Human Genome U133 Plus 2.0 Array (Affymetrix)
- GeneChip Mouse Genome 430 2.0 Array (Affymetrix)
- Whole Human Genome Microarray 4x44K v2 (Agilent)
- Whole Mouse Genome Microarray 4x44K v2 (Agilent)
のデータを取り込んでおり、随時拡充していく予定です。
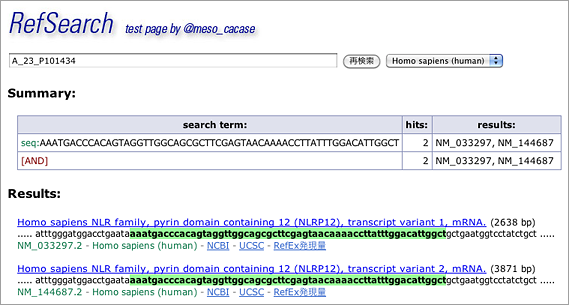
下記はAgilentのヒトのアレイ(Whole Human Genome Microarray 4x44K v2)から A_23_P101434 を検索した例(リンク)。Agilentのアレイは基本的には60-merのオリゴ1本で1つのtranscriptを認識するようにできている点がAffymetrixとの大きな違いです。mesoの経験的にはこちらのほうがバックが小さくなり、発現量が比較的少ない遺伝子まで綺麗にデータが出るようです。
- Comments (Close): 0
- Trackbacks: 0
Home > Archives > 2011-06
- Search
- Feeds
- Meta