Home > Archives > 2011-10
2011-10
トーゴーの日シンポジウム2011
- 2011-10-11 (火)
- DBCLS
10月5日(水) に日本科学未来館で開催された「トーゴー(統合)の日シンポジウム2011」でGGRNAの紹介をしました。
ポスター会場ではiPadを使ってGGRNAのデモをしましたが、やはり検索が速いという感想を多くいただけました。また、疾患関連のキーワードで遺伝子を検索したいという要望や、SNP、タンパクのモチーフを見たいという要望を頂いており、これらはGGRNAに取り入れていく方針です。(疾患関係は翌日の某ユーザ会でも話題になりました。)
ポスターのPDFと要旨を下記に掲載します。
・ポスターPDF (1.7MB)
「統合遺伝子検索GGRNA」(ポスター番号 13)
〇内藤雄樹,坊農秀雅(情報・システム研究機構 ライフサイエンス統合データベースセンター)
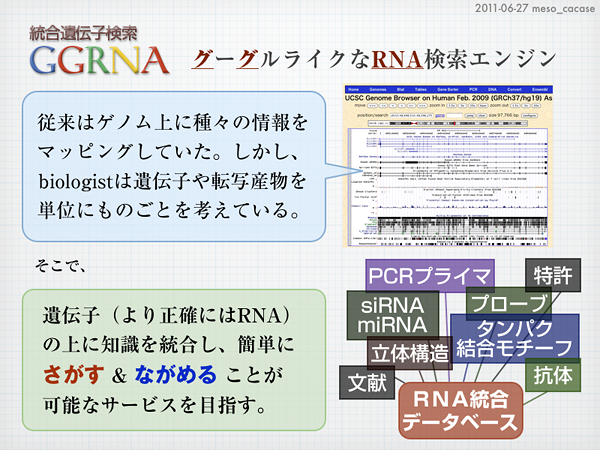
要旨:さまざまな切り口で遺伝子を「さがす」または「ながめる」ことができるように,塩基配列,発現情報,立体構造,文献等さまざまな知識をRNA配列(転写産物)をベースに整理した「RNA統合データベース」を構築している.その一環として,RefSeqに収録された遺伝子を簡便かつ高速に検索できるウェブサービスGGRNA(http://GGRNA.dbcls.jp/)を構築したので紹介する.GGRNAは,単一の検索窓しかないシンプルなインターフェースから,RefSeqに収録された転写産物の各種ID,遺伝子名,アノテーション情報,塩基配列およびアミノ酸配列を含む全フィールドを対象に全文検索をおこなうシステムである.とくに塩基配列の検索では,Nなどの曖昧な塩基を含むパターンや,クエリに対して2塩基までのミスマッチを含む配列を高速に検索する.またマイクロアレイのプローブIDを入力すると,そのプローブの塩基配列をもとに遺伝子を検索することもできる.現在,ヒト,マウス,ラット,ニワトリ,ツメガエル,ゼブラフィッシュ,カタユウレイボヤ,ショウジョウバエ,線虫,イネ,シロイヌナズナ,出芽酵母,分裂酵母に対応している.
- Comments (Close): 0
- Trackbacks: 0
GGRNAの「統合検索」始動
- 2011-10-07 (金)
- DBCLS
外部コンテンツと連携して「統合検索」
今回、GGRNAを公開してから最大のアップデートをおこないました。
GGRNAのトップページをよく見ると「統合遺伝子検索」と銘打っているわけですが、今までのGGRNAは「RefSeqの全文検索」以上のものではありませんでした。では、「統合・・・」とはどういうことなのか。mesoが目指しているのは、様々なコンテンツを(ゲノムではなく)転写産物に紐付けて「さがす」&「ながめる」ことができる仕組みです。
今回のアップデートでは、GGRNA内部でデータを保持する方法を書きかえ、RefSeq以外のコンテンツを転写産物に紐付けて扱えるようにしています。手始めにGene Ontologyによるアノテーション情報と、酵素EC番号の情報とを、それぞれNCBI Entrez Geneから抽出してRefSeqのtranscriptに統合、GGRNAで検索できるようにしました。
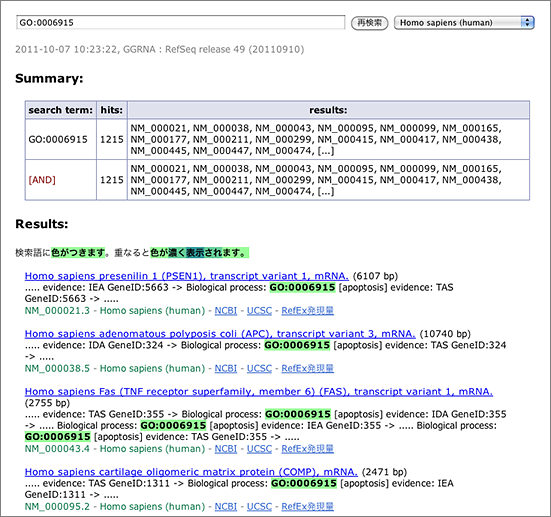
Gene Ontologyで検索
アポトーシス関連遺伝子を表すGene OntologyのID、GO:0006915 で検索(→ GGRNA検索)。
ヒットした転写産物をクリックします。各転写産物に紐付けられた情報を見ることができます。
GGRNAは、実はこのテキストを全文検索して GO:0006915 という部分を見つけているだけなので、[apoptosis] で検索してもOK(→ GGRNA検索)。上記の GO:0006915 で検索した場合と同じ件数がヒットします。括弧をつけずに apoptosis で検索すると、Gene Ontology情報以外のところに apoptosis という単語が含まれるものが全部ヒットしてしまうので4000件以上になります。
それでは困るという場合、「転写産物に紐付けされた外部コンテンツ」のみを対象に検索するには anot: タグ(annot: , annotation: と書いてもよい)を使います(e.g. anot:GO:0006915 → GGRNA検索)。ただし、anot:apoptosis で検索した場合(→ GGRNA検索)は GO:0006915 [apoptosis] だけでなく、GO:0006916 [anti-apoptosis] とか GO:0042981 [regulation of apoptosis] もヒットします。
なお、スペースを含む場合はダブルクオートで囲みます(e.g. anot:“regulation of apoptosis” → GGRNA検索)。
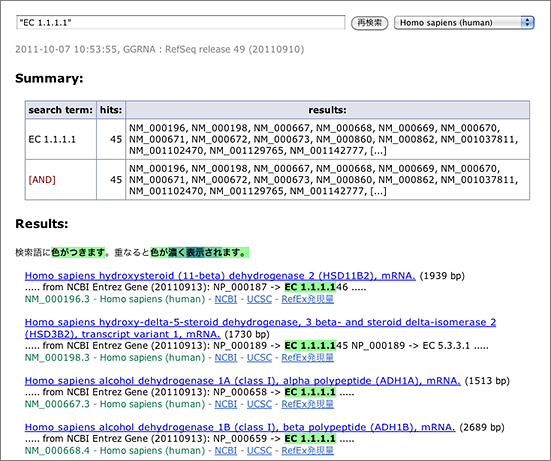
酵素EC番号で検索
アルコールデヒドロゲナーゼ、EC 1.1.1.1 で検索(→ GGRNA検索)。anot: タグを使う場合はECの後にスペースがあるのでダブルクオートで囲み、anot:“EC 1.1.1.1” とします(→ GGRNA検索)。
EC 1.1.1.1 だけでなく、EC 1.1.1.146 や EC 1.1.1.145 がヒットしているのはご愛敬。何とかします。
なお、Gene Ontologyによるアノテーションや酵素EC番号以外にも、様々なコンテンツを今後統合していく予定です。10/5に行われたトーゴーの日シンポジウム2011ではGGRNAで疾患関連の情報やSNP、タンパクのモチーフなどを見たいという要望も頂いており、さっそく検討しています。
トップページURLを変更
トップページを http://GGRNA.dbcls.jp/ に変更し、このURLで本格運用することにしました。GGRNAの部分は小文字でも可。一般的にドメイン名は大文字小文字を区別しないルールになっています。なお、旧アドレスは新アドレスに転送されます。
RefSeq 49にアップデート、ホヤを追加
RefSeq Release 49 (Sep, 2011) にアップデートしました。同時に @wakuteka さんのリクエストでホヤCiona intestinalisを追加。ただし現時点では情報が充実していないようで、NM_* が901件に対して XM_* (predicted) が13041件でした。RefSeqのヘルプによると、RefSeq ID(アクセッション番号)の先頭が NM_ のものは「Transcript products; mature messenger RNA (mRNA) transcripts」で、XM_ のものが「Transcript products; model mRNA provided by a genome annotation process; sequence corresponds to the genomic contig」となっています。つまり XM_ のほうはRNAの分子としてまだ確認されていないもの、という言い方ができると思います。
塩基配列検索を強化
3ミスマッチまで許容して塩基配列を検索できる seq3: というタグを用意しました。相補鎖を3ミスマッチまで検索する comp3: 、両方の鎖を3ミスマッチまで検索する both3: も使えます。ただし短い配列を検索すると非常にたくさんの配列がヒットして時間がかかるので17塩基以上を推奨します。
ちなみに「caagaagagattgtg」という15塩基の配列を、完全一致検索、1ミスマッチ、2ミスマッチ、3ミスマッチまで検索、としていくとヒット件数は約10倍ずつ増えていきます。
- 「caagaagagattgtg」で検索(→ GGRNA検索)….. 2件
- 「seq1:caagaagagattgtg」で検索(→ GGRNA検索)….. 28件
- 「seq2:caagaagagattgtg」で検索(→ GGRNA検索)….. 365件
- 「seq3:caagaagagattgtg」で検索(→ GGRNA検索)….. 3522件
なお、GGRNAでは他にもいろいろな検索タグを用意しています。目的に応じて利用すれば効率的に検索できるでしょう。検索タグの一覧はヘルプのページにあります。
- GGRNAヘルプ — http://GGRNA.dbcls.jp/help.html
- Comments (Close): 0
- Trackbacks: 0
Home > Archives > 2011-10
- Search
- Feeds
- Meta