Home > Archives > 2013-08
2013-08
GGRNA ver.2リリース
- 2013-08-23 (金)
- DBCLS
7月8日に GGRNA ver.2 をリリースしました。GGRNAを公開してから初のメジャーアップデートになります。少し時間が経ってしまいましたが、この記事では、アップデートによる主な変更点をまとめました。
全生物種に拡張
これまでのGGRNAは、RefSeqに収録された転写産物のうち、13種類のモデル生物に限ってサービスを提供してきました。今回のアップデートでは、これを全生物種に拡張し、しかも同時に検索できる Zoo というモードを用意しました(検索例:CAAGAAGAGATTG )。
ちなみに RefSeq は、ゲノム、mRNA、ncRNA、タンパク質のエントリから構成されており、GGRNAではこのうちmRNA(IDがNM_, XM_で始まる)および ncRNA(NR_, XR_)を検索できます。
GGRNAの元データ(NCBI RefSeq)
ftp://ftp.ncbi.nlm.nih.gov/refseq/release/complete/complete.*.rna.gbff.gz
技術面では、データベースの増大に対応するために、内部の検索エンジンを最新版の Sedue に置き換えました。これまでのGGRNAでは、圧縮接尾辞配列のインデックスをメモリに載せて検索していたため、メモリ容量の制約で13種類のモデル生物しか入りませんでした。また、ヒット件数が極端に多い場合に、圧縮接尾辞配列の解凍処理がネックになりすごく時間がかかるという弱点がありました。最新版のSedueでは(非圧縮の)接尾辞配列のインデックスをSSDに載せることによって、これらの弱点を克服しています。
最近SSDはずいぶん安価になってきて、2013/8/20の時点で960GBのモデルが5.9万円で販売されています(価格.com)。約3TBのスペースを確保すれば、GenBank/EMBL/DDBJ国際塩基配列データベースに登録されたゲノム以外の全てのエントリを、配列を含めて全文検索できるようになります。実はもう手元のテスト環境では動いており、データ更新などいくつかの問題を解決できたら公開する予定です。
REST APIを完備
クエリとURIとを1対1対応させました。検索結果に直接リンクを張ったり、機械的にアクセスして検索結果をテキスト形式やJSONで取得することが、より簡単にできるようになりました。
URI
http://GGRNA.dbcls.jp/species/query+string[.format][.download]species → 生物種。hs (ヒト), mm (マウス), dm (ハエ), …。省略時はヒト。
query+string → 検索ワード。特殊文字はURIエンコードすること。
format → html, txt, json。省略時はhtml。
download → ファイルとしてダウンロード。詳細は GGRNA.v2 REST API仕様 を参照。
検索例:
- http://GGRNA.dbcls.jp/NM_001518
→「NM_001518」を検索。.format の部分を省略した場合は html になる。 - http://GGRNA.dbcls.jp/hs/homeobox.txt
→ ヒトで「homeobox」を検索してタブ区切りテキストで取得。 - http://GGRNA.dbcls.jp/dm/%22RNA+interference%22.json
→ ショウジョウバエで「”RNA interference”」をフレーズ検索してJSONで取得。
URIでは ” は %22、スペースは + に置換する(URIエンコード)。 - http://GGRNA.dbcls.jp/ce/caagaagagattg.txt.download
→ 線虫で「caagaagagattg」を検索してタブ区切りテキストをダウンロード。
なお、旧バージョンで提供してきたREST APIもそのまま残しており、以前と同じURIで情報を取得できますが、
http://GGRNA.dbcls.jp/api/* → http://GGRNA.dbcls.jp/v1/api/*
にリダイレクトされる点はご注意ください。
マイクロアレイのプローブ情報を網羅的に収録
GGRNAでマイクロアレイのプローブIDを検索すると(例:1552311_a_at )、自動的にプローブの配列に変換したうえでその結合部位を検索する機能があります。今回のアップデートでGGRNAはあらゆる生物種に対応したので、それに合わせてマイクロアレイのプローブ情報も追加しました。詳細は別の記事に書きますが、AffymetrixとAgilentのほぼ全てのマイクロアレイ製品のプローブIDと配列を網羅的に収録しています。
検索結果のランキングを改良
遺伝子名のつもりで Dicerを検索 しても、アミノ酸配列のなかの “DICER” ばかり上位に表示されて残念だったので、キーワードがヒットした場所に応じて重みを付けることにしました。具体的には、遺伝子名やタイトルにマッチした場合は相対的に高いスコアを、アミノ酸配列や塩基配列にマッチした場合は相対的に低いスコアを与えています。
ランキングは今後さらに改良していく必要があり、たとえばメジャーな生物種をより上位に表示したり、Accession番号が XM_/XR_ で始まる “PREDICTED” な配列のスコアを相対的に下げたりすると使いやすくなりそうです。ただし、なるべく多くのユーザの意図に沿うバランスの良いスコアリングを慎重に検討する必要があります。また、GGRNAのランキングがユーザの意図に沿っていなかった場合でも(たとえば本当に “DICER” というアミノ酸配列を探したかった場合でも)、ファセットナビゲーションのような仕組みで即座に目的のものを絞り込めるようにしたいです。
塩基配列のミスマッチ検索を廃止 → GGGenomeに移行
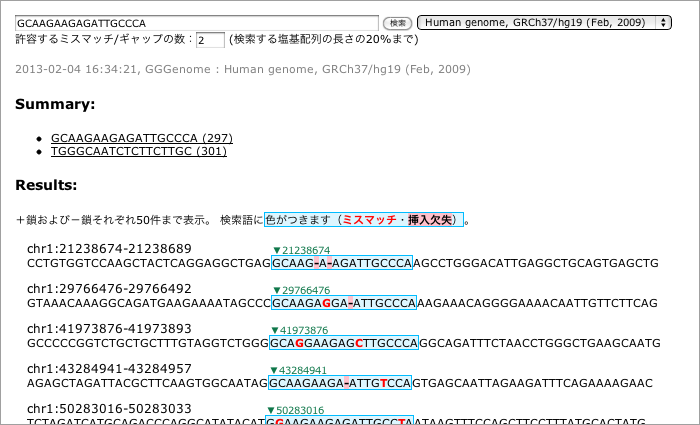
GGRNAの内部エンジンを変更したことにより、ミスマッチを含む塩基配列の検索ができなくなりました。配列検索にこだわってきたGGRNAとしては、このダウングレードは痛手なのですが、データベースの拡張性を優先してミスマッチ検索の廃止を決めました。そのかわりに、塩基配列の検索に特化した GGGenome《ゲゲゲノム》というサイトを新たに立ち上げ、ミスマッチやギャップを含む塩基配列の検索はそちらに引き継ぐことにしました。
高速塩基配列検索 GGGenome《ゲゲゲノム》
http://GGGenome.dbcls.jp/
GGGenomeでミスマッチやギャップを含む塩基配列を検索した例:
Gene OntologyおよびEC番号検索の停止
旧バージョンでは、Gene OntologyのIDやterm、酵素EC番号の情報をRefSeqの転写産物に紐付けることによって、GGRNAで検索できるようにしていました。しかし、この機能も内部エンジンの更新に伴いしばらく提供できなくなります。こちらは再開に向けて開発を進めます。
ソースコード公開
今回のアップデートを機にソースコードを公開しました。
ソースコード(GitHub)
https://github.com/meso-cacase/GGRNA
内部エンジンに使用している Sedue は商用ソフトウェアでソースは公開されていませんが、プリファードインフラストラクチャー(PFI)から入手が可能です。Sedueでは、接尾辞配列のインデックスをSSDに保持することによって、塩基配列やアミノ酸配列を含む検索ワードを、見落としなく、きわめて高速に検索しています。
旧バージョン
今回のアップデートでは大きく変更した部分や削除した機能もあるので、旧バージョンもしばらく提供します。ただし、RefSeqのリリースに合わせた2ヶ月に一度のデータベース更新はせず、2013年7月のRefSeq release 60で更新を終了します。
GGRNA ver.1
http://GGRNA.dbcls.jp/v1/
- Comments (Close): 0
- Trackbacks: 0
Home > Archives > 2013-08
- Search
- Feeds
- Meta