Home > Archives > 2014-08
2014-08
マイクロアレイ学生実習2014
- 2014-08-01 (金)
- DBCLS
6/10〜18の期間、東京大学理学部生物化学科・生物情報科学科3年生の学生実習を担当しました。テーマは「マイクロアレイを用いた網羅的遺伝子発現解析」です。生化・生物情報では、2009年にアジレント社のマイクロアレイ設備一式を学生実習のために導入しており、学科の共通機器として毎年の学生実習に使用しているほか、各ラボの研究にも活用されています。
siRNAのオフターゲット効果をゲノムワイドに調べる
今年の学生実習では、マイクロアレイを用いてsiRNAのオフターゲット効果をゲノムワイドに調べる実験を行いました。 siRNAは、細胞内において本来の標的とは無関係なmRNAと結合して意図せず抑制してしまうことがあり、そのような現象をsiRNAの オフターゲット効果 と呼びます。オフターゲット効果は、siRNAを用いた解析で誤った解釈を導く原因となるほか、siRNAを核酸医薬品として応用する際に副作用の原因となるなど、RNAi法における深刻な課題のひとつと考えられます。このため、どのような場合にオフターゲット効果がおこるのか、siRNAとmRNAとのあいだの配列類似性に着目し、さまざまな解析がなされてきました。その結果、siRNAの5′末端から2-8の位置の ‘seed’ と呼ばれる7塩基の領域がmRNAの3′ UTRと相補的な場合に、mRNAの分解や翻訳抑制が起こり得ることがわかってきました (Lim et al., 2005; Lin et al., 2005; Birmingham et al., 2006)。
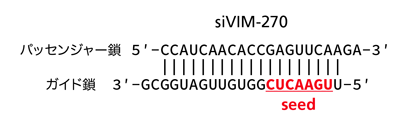
たとえば、上図は今回の実習で使用したヒトのビメンチン (NM_003380) を標的とするsiRNAで、ガイド鎖の配列のうち赤字の 5′-UGAACUC-3′ がseedと呼ばれる領域です。前述のように、これと相補的な 5′-GAGUUCA-3′ という配列を3′ UTRにもつ (以下 seedマッチする と表現) mRNAは、オフターゲット効果により抑制される場合があります。実習では、その様子をマイクロアレイでゲノムワイドに検証していきます。さらに、通常のsiRNAだけでなく、一部を化学修飾したsiRNAを4種類用意し、それらのオフターゲット効果を通常のsiRNAと比較します。本来の標的遺伝子であるビメンチンを十分に抑制し、一方でオフターゲット効果の少ない化学修飾siRNAが見つかれば、有望かもしれません。
マイクロアレイ実験およびデータ解析
実習では、上記のsiRNAをHeLa細胞に導入して24時間後に回収したものを各班に配布し、1日目にtotal RNA抽出、2日目に逆転写とcRNA合成、3日目にアレイへのハイブリダイゼーション、4日目にアレイのスキャンをおこない、4〜6日目の3日間でデータ解析をしました。データ解析の3日間は情報基盤センターの演習室をお借りし、iMac端末で下記のようなソフトウェアを使用して解析を行いました。
- Excel – マイクロアレイのデータ解析をブラックボックスにしないために、まずは生の数値にきちんと触れてもらいました。サンプル間の正規化を行い各種プロットを作成。
- GeneSpring GX – エクセルで全部やるのは苦行なので、一連のデータ解析は基本的にGeneSpring GXまたはRを使用。GeneSpring GXはアジレント社より学生実習のための期間限定ライセンスを供与いただきました。
- R – マイクロアレイデータ解析用のパッケージlimmaを利用し、コードの基本部分を講師が提供。
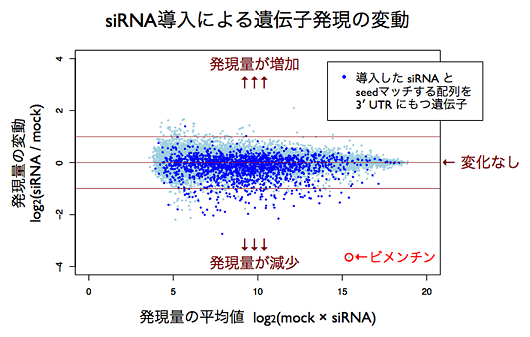
細胞に導入したsiRNAとseedマッチする遺伝子をリストアップして、それらの発現量の変動をMAプロットにより可視化してみます。すると、seedマッチする遺伝子群 (青色) は、その他の遺伝子群 (水色) と比較して下側に分布しており、発現量が減少している傾向が確認できました。ただし減少といってもあくまで集団としての傾向で、個別の遺伝子に注目すると、seedマッチしていても変化がなかったり、増加していたりする遺伝子もたくさんあります。また、本来の標的であるビメンチン (下図の赤丸) は -3.6 程度であり、log2をとる前の値に直すと 8% 程度に抑制されていることが確認できました。
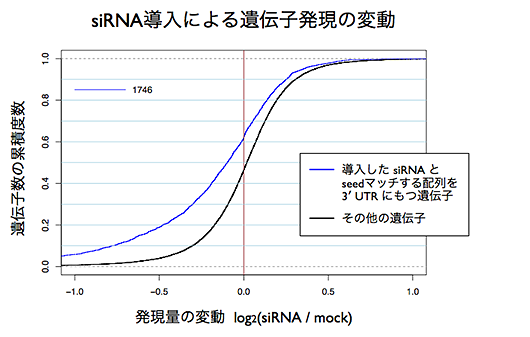
発現量が減少している傾向を、より定量的に評価してみます。seedマッチする遺伝子群 (上のMAプロットで青色)、その他の遺伝子群 (水色) のそれぞれに対し、発現量の変動 (MAプロットの縦軸の値) でソートしたときの累積度数を求め、プロットしました。
コントロールである その他の遺伝子群 (黒色の線) は、概ね (0, 0.5) を通る対称的な曲線になりました。一方、seedマッチする遺伝子群 (青色の線) は、それよりも左方向にシフトしており、発現量が減少している傾向がはっきりとわかりました。もしまったく減少していなければ、黒い曲線と重なるはずです。このように、累積度数をプロットすると、MAプロットでは減少がわかりにくい場合でも、定量的に評価できるようになります。
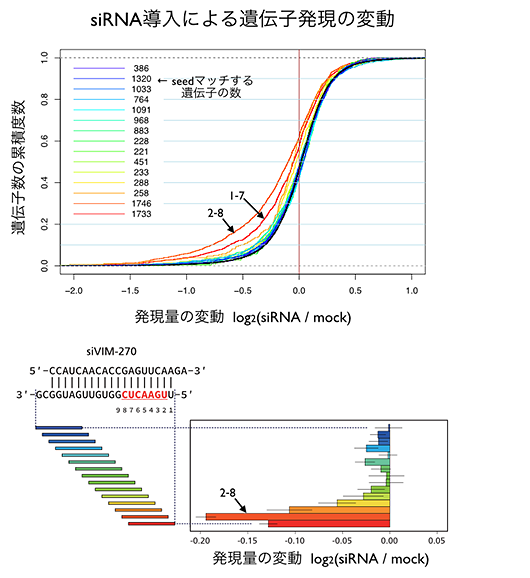
ところで、seedの2-8という位置が抑制に寄与することは、もともとモチーフ解析などから明らかになってきたものですが、それ以外の位置の7mer (1-7, 3-9, 4-10, …) は抑制に寄与しないのでしょうか。このことを調べるために、前述の累積度数プロットを、1-7の配列 (UUGAACU) とマッチする遺伝子群 (赤線)、2-8の配列 (UGAACUC) とマッチする遺伝子群 (朱色)、3-9の配列 (GAACUCG) とマッチする遺伝子群 (橙色)、…(以下同様)… それぞれについて描画しました (下図)。
この図から、朱色でプロットされた2-8の配列 (seed) とマッチする遺伝子群が、もっとも抑制されることが確認できました。さらに見やすくするために、黒色の線と囲まれた部分の面積、つまり曲線がどれだけ左にシフトしたかを求め、棒グラフで示しました。2-8の配列 (seed) とマッチしている遺伝子群がもっとも抑制されていますが、1-7や3-9の配列とマッチしている遺伝子群も、ある程度は抑制される傾向があることがわかりました。
さらに実習では、siRNAの反対側のstrandのseedがマッチした場合や、化学修飾したsiRNAのオフターゲット効果についても同様に解析しました。それらの結果はここでは割愛しますが、6日間の学生実習でこのように様々な解析ができたことは、よかったのではないかと思います。
- Comments (Close): 0
- Trackbacks: 0
Home > Archives > 2014-08
- Search
- Feeds
- Meta