- 2011-07-29 (金) 3:11
- DBCLS
Nを含む塩基配列の検索
GGRNAで塩基配列を検索する場合、今まで seq1: や seq2: のタグでそれぞれ1塩基または2塩基以内のミスマッチを許容する検索ができましたが、AGGTCANNNTGACCT のようにあいまいな塩基を含む検索には対応していませんでした。今回のアップデートでは、iub:AGGTCANNNTGACCT(→GGRNAで検索)とすることで、あいまいな塩基(IUBコード)を含む配列を検索できるようにしました。狙いは、RNA結合タンパクが認識するあいまいなモチーフを検索したり(例:PUM結合サイト iub:UGUANAUA →GGRNAで検索;10秒くらいかかる)、GGRNAのエンジンをゲノム検索に応用して転写因子結合サイトなどを探すことを想定しています。(そのためには、もっとタンパクが核酸とくっつく気持ちで配列検索できるようなプログラムを書きたい・・・)
(参考:IUBコード表)
| Code | Base | Comment |
|---|---|---|
| R | A, G | puRines |
| Y | C, T | pYrimidines |
| M | A, C | aMino |
| K | G, T | Keto |
| S | C, G | Strong |
| W | A, T | Weak |
| H | A, C, T | not G |
| B | C, G, T | not A |
| V | A, C, G | not T |
| D | A, G, T | not C |
| N | A, C, G, T | aNy |
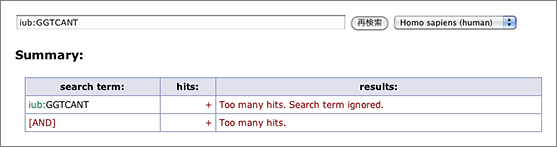
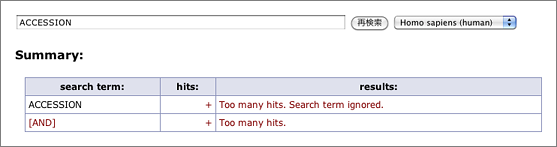
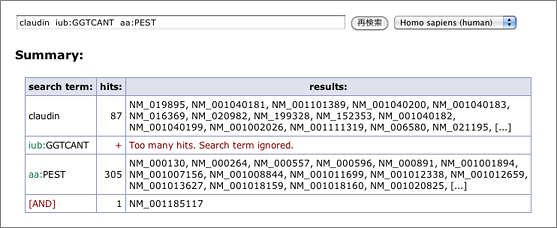
N等のあいまいな塩基が多くなるほどヒット件数が爆発的に増えて検索に時間がかかります。そこで今回のアップデートでは、一つのキーワードがヒットするRefSeq IDの件数が10,000を越える場合、そのキーワードは「絞り込みに寄与しない」と見なして無視するようにしました。具体的に説明すると、たとえば iub:GGTCANT では15,000件以上のRefSeq IDがヒットします。また ACCESSION で検索すると、(RefSeqのデータには必ずACCESSIONという文字列が含まれているので)今までは全件がヒットしていました(泣)。今後は、このようなキーワードを単独で検索すると “Too many hits.” として何も表示せず、またAND検索ではそのキーワードを含めずに検索した結果を返すことにしました。
(iub:GGTCANT で検索 → “Too many hits.”)
(ACCESSION で検索 → “Too many hits.”)
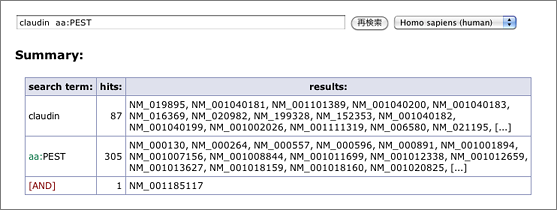
(claudin iub:GGTCANT aa:PEST で検索 → claudin aa:PEST で検索した場合と同じ結果)
ツメガエル、イネの追加
Twitterなどで要望をいただいたツメガエルXenopus (Silurana) tropicalisとイネOrysa sativaを追加。ただし中身を眺めてみると、ツメガエルはRefSeq IDがXM_,XR_で始まる “PREDICTED: ~” な遺伝子のほうが多いし(23,899件中15,110件)、イネは遺伝子名がついておらずsymbolがすべてOs02g0617400のような名前になっています。今後のRefSeqのアップデートに期待したいです。
RefSeqアップデート
Release 48 (Jul, 2011) にアップデートしました。RefSeqは2ヶ月に1回のペースでリリースされるので、更新の作業もだいぶ自動化しました。余談ですが、データをダウンロードするのにlftpのpgetを利用すると、1つのファイルを分割して各パートを並列ダウンロードするのでかなり速くなります。
lftp -c "mirror -i rna.gbff --parallel=3 --use-pget-n=3 \
ftp://ftp.ncbi.nlm.nih.gov/refseq/release/complete RefSeq_DL/"
- 「mirror」はディレクトリをまるごとミラーするlftpのコマンド。
- 「-i rna.gbff」はファイル名に rna.gbff を含むものだけをダウンロード。
- 「–parallel=3」は3ファイルを並列ダウンロード。
- 「–use-pget-n=3」は1つのファイルを3分割して並列ダウンロード。
マイクロアレイのプローブ情報を追加
ツメガエルとイネのデータを追加したことに伴い、以下のアレイのプローブ情報を追加しました。
| メーカー | 生物種 | マイクロアレイの種類 | GPL ID |
|---|---|---|---|
| Affymetrix | ツメガエル | Xenopus laevis Genome Array | GPL1318 |
| Affymetrix | ツメガエル | Xenopus laevis Genome 2.0 Array | GPL10756 |
| Affymetrix | ツメガエル | Xenopus tropicalis Genome Array | |
| Affymetrix | イネ | Rice Genome Array | |
| Agilent | ツメガエル | X. laevis (Frog) Oligo Microarray v2 (023448) | |
| Agilent | ツメガエル | Xenopus laevis (Frog) Oligo Microarray (013665) | |
| Agilent | ツメガエル | Xenopus Gene Expression Microarray (015066) | GPL11258 |
| Agilent | イネ | O. sativa (Rice) Oligo Microarray 4x44K (015058) | |
| Agilent | イネ | Rice Gene Expression Microarray (015241) | GPL6864,GPL8852 |
| Agilent | イネ | Rice Microarray (012106) | GPL892,GPL7268 |
今後の方針もろもろ
昨日、@h_ono さんの出身ラボの方々からGGRNAについてフィードバックを頂きました。インターフェースで改善すべき点からGGRNA自体の位置付けまで、ウェットな現場からの辛口な、しかしどれも納得のご指摘でした。やはり現場で「使える」ものでなければ作る意味がないわけで、あくまで生物学を解くことが開発の目的であるということを忘れないようにしたい。それを踏まえ、GGRNAが生命科学研究にどう使えるのかを、NCBIのサイトではできないことを中心に次回紹介したいと思います。
- Newer: GGRNA活用事例集(塩基配列編)
- Older: 統合データベース講習会 AJACS本郷9
Trackbacks:0
- Trackback URL for this entry
- /archives/899/trackback
- Listed below are links to weblogs that reference
- Nを含む塩基配列の検索、DB追加+アップデート from mesoの実験ノート