- 2014-08-01 (金) 15:46
哺乳類細胞で有効かつ標的遺伝子に特異的なsiRNA設計法の確立とその抗ウイルスRNA干渉法への応用
Antiviral RNA interference based on the development of the highly effective, target-specific siRNA design in mammalian cells
平成18年12月 博士(理学)申請
東京大学大学院理学系研究科 生物化学専攻 内藤 雄樹
・学位論文 (1) – 要旨・緒論
・学位論文 (2) – 第一章(本記事)
・学位論文 (3) – 第二章
・学位論文 (4) – 文献・巻末表・謝辞
第一章 哺乳類細胞で有効かつ標的遺伝子に特異的なsiRNA設計法の構築
1. 結果と考察
1-1. 哺乳類細胞で活性の高いsiRNA設計法の検討
哺乳類細胞では,無作為に設計したsiRNAのうち2〜3割程度しか十分なRNAi活性を示さず,それ以外の多くのsiRNAは機能しないことが知られている(Holen et al., 2002).この問題に対してUi-Teiおよび著者らは,哺乳類細胞で高いRNAi活性を示すsiRNAの配列規則性を見いだした(Ui-Tei et al., 2004).また,他の研究グループからも,哺乳類細胞で有効なsiRNAの設計法について,実験結果に基づく具体的な方法が提唱されている(Reynolds et al., 2004; Amarzguioui et al., 2004).本研究ではまず,Ui-Teiら,Reynoldsら,Amarzguiouiらの3つのsiRNA設計法の比較評価をおこなった.
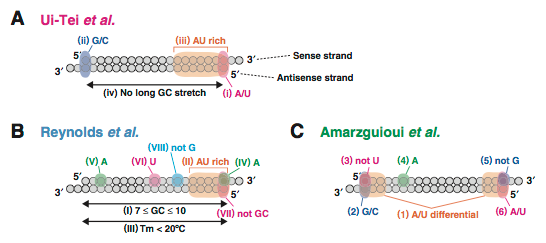
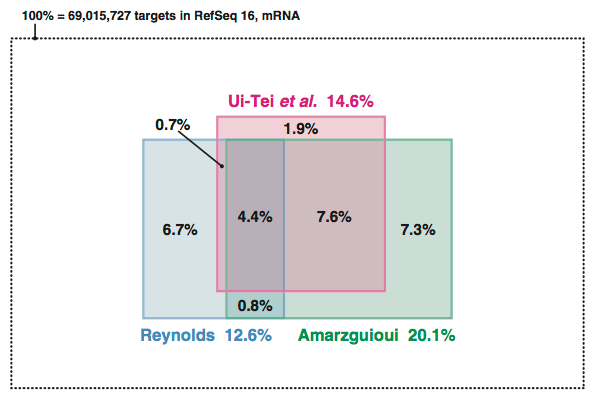
Ui-Teiら,Reynoldsら,Amarzguiouiらの各siRNA設計法は,いずれもsiRNAの配列自体からそのRNAi活性を予測し,有効なsiRNAを選択しようとするものである(Fig. 1-1).まず,siRNAを無作為に設計した場合に,それぞれの設計法を満たすsiRNAがどのような割合で設計できるかを検討した.RefSeq(Pruitt et al., 2007)release 16に登録されたヒトの全遺伝子に対して設計可能な69,015,727種のsiRNAのうち,Ui-Teiら,Reynoldsら,Amarzguiouiらの設計法を満たすsiRNAは,それぞれ14.6%,12.6%,20.1%であった(Fig. 1-2).各siRNA設計法は,いくつかの相違点があるものの,siRNAを構成するアンチセンス鎖の5′末端がAまたはUであることが好ましく,アンチセンス鎖の5′末端からある程度の長さ(3〜7塩基)にAまたはUが多いほうが有効である点で一致している.このため各設計法を満たすsiRNAの集合は互いに重なり合い,3種の設計法をすべて満たすsiRNAも全体の4.4%存在した.また特に,Ui-Teiらの設計法およびAmarzguiouiらの設計法は,センス鎖の5′末端がGまたはCであることが好ましいという点も共通であるため,互いによく重なり合った.
Figure 1-1. 哺乳類細胞で有効なsiRNAの設計法.(A) Ui-Teiらの提唱するsiRNA設計ガイドライン(Ui-Tei et al., 2004).(i)〜(iv)の条件をすべて満たすsiRNAを有効と予測している.(B) ReynoldsらのsiRNA設計法(Reynolds et al., 2004).(I)〜(VIII)の条件をできるだけ満たすsiRNAを有効と予測している.(C) AmarzguiouiらのsiRNA設計法(Amarzguioui et al., 2004).(1)〜(6)の条件をできるだけ満たすsiRNAを有効と予測している.
Figure 1-2. ヒトの全遺伝子に対して設計可能なsiRNAの全体を100%として,Ui-Teiら,Reynoldsら,Amarzguiouiらの設計法を満たすsiRNAの割合をそれぞれ示した.
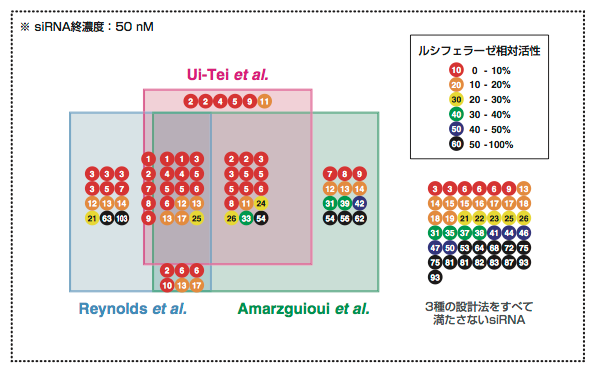
各siRNA設計法の有効性を検証するため,ホタルルシフェラーゼ遺伝子を標的とする114種のsiRNA(Table S1, 巻末)のRNAi活性を,ルシフェラーゼアッセイにより評価した.114種のsiRNAの内訳は下記のとおりである.なお,詳細はTable S1(巻末)に示した.
-
3種の設計法をすべて満たさないsiRNA 43種
-
3種の設計法のうち1種のみを満たすsiRNA 30種
-
3種の設計法のうち2種を満たすsiRNA 26種
-
3種の設計法をすべて満たすsiRNA 15種
これらのsiRNAのRNAi活性を,ルシフェラーゼアッセイで評価した結果をTable S1(巻末)およびFigure 1-3に示した.Ui-Teiら,Reynoldsら,Amarzguiouiらの各設計法を満たすsiRNAのうち,ルシフェラーゼ相対活性を50%以下に抑制したsiRNAは,それぞれ
-
Ui-Teiらの設計法 40 / 41 (97.6%)
-
Reynoldsらの設計法 36 / 38 (94.7%)
-
Amarzguiouiらの設計法 44 / 48 (91.7%)
であった.また,ルシフェラーゼ相対活性を10%以下に抑制した,高いRNAi活性を有するsiRNAは,
-
Ui-Teiらの設計法 30 / 41 (73.2%)
-
Reynoldsらの設計法 25 / 38 (65.8%)
-
Amarzguiouiらの設計法 27 / 48 (56.3%)
であった.3種の設計法とも哺乳類細胞で有効なsiRNAを効率よく選択できたが,特にUi-Teiらの設計法が良好な成績であった.また,Reynoldsらの設計法のみを満たす12種のsiRNAと,Amarzguiouiらの設計法のみを満たす12種のsiRNAとに注目すると,RNAi活性が低いsiRNAが相対的に多いことが判った(Fig. 1-3).一方,Ui-Teiらの設計法のみを満たす6種のsiRNAは,いずれも有効であった(Fig. 1-3).Ui-Teiらの設計法は,他の設計法よりも判定項目が少なく単純であるが,すべての判定項目を満たすsiRNAのみを有効としている点でReynoldsらやAmarzguiouiらの設計法と異なっており,このため有効なsiRNAを効率よく選択できたのかも知れない.本研究では以上の結果を考慮し,Ui-Teiらの設計法のみを採用してsiRNA設計をおこなうこととした.
Figure 1-3. ホタルルシフェラーゼ遺伝子を標的とする114種のsiRNAを,Ui-Teiら,Reynoldsら,Amarzguiouiらの各設計法への適合性に基づき分類し,RNAi活性とともに示した.ルシフェラーゼ相対活性の値が低いほど,siRNAのRNAi活性が高いことを示している.
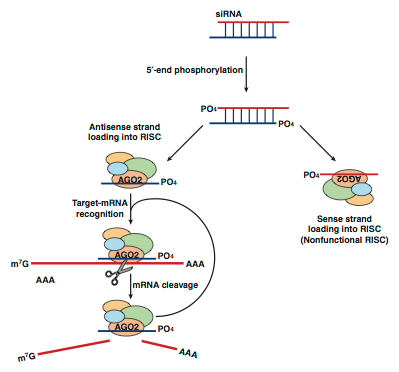
なお,Ui-Teiらの設計法によって有効なsiRNAを選択できる理由は以下のように考えられる.RNAiの実行過程において,siRNAは複数のタンパク質と複合体(RISC)を形成するが,このときRISCにはsiRNAを構成する2本のRNA鎖のうち1本のみが取り込まれる(Martinez et al., 2002).siRNAのアンチセンス鎖側がRISCに取り込まれた場合,RISCは標的mRNAを認識して切断できるが,センス鎖側がRISCに取り込まれた場合は標的mRNAを認識できず,mRNAは切断されない(Fig. 1-4).Schwarzらは,ショウジョウバエのin vitroの実験系を用いて,siRNAのどちらのRNA鎖がRISCに取り込まれるかを,siRNAの両末端部分の塩基対形成の強さに注目して解析した.その結果,siRNAのアンチセンス鎖の5′末端側が,センス鎖の5′末端側よりも塩基対形成が弱い場合に,siRNAのアンチセンス鎖がおもにRISCに取り込まれていることが判明した(Schwarz et al., 2003).またKhvorovaらは,ヒト培養細胞においてRNAi活性が高いsiRNAは,アンチセンス鎖の5′末端側の塩基対が熱力学的に安定性が低くなっており,このことがRISCにどちらのRNA鎖が取り込まれるかを規定すると報告している(Khvorova et al., 2003).これらの報告は,Ui-Teiらの設計法の,アンチセンス鎖の5′末端およびその周辺にAまたはUが多く,センス鎖の5′末端がGまたはCであるようなsiRNAが有効であるという条件と対応している.ただし,siFL-1063(Table S1; 巻末)のように,siRNAのアンチセンス鎖の5′末端から9塩基が連続してGまたはCであっても活性の高いsiRNAが存在することから,末端部分の塩基対の安定性以外にも,RISC形成におけるRNA鎖の選択に関わる要因が存在すると考えられる.
Figure 1-4. RISCにはsiRNAを構成する2本のRNA鎖のうち1本のみが取り込まれる.siRNAのアンチセンス鎖がRISCに取り込まれた場合は標的mRNAを認識して切断できるが,センス鎖が取り込まれた場合は標的mRNAを認識できない.
1-2. ゲノムワイドなsiRNA設計への適用可能性
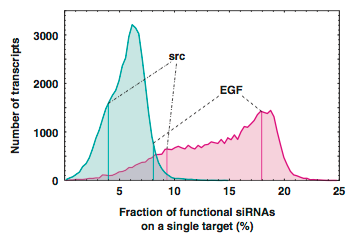
Ui-Teiらの設計法では,ヒトの全遺伝子に対して設計可能なすべてのsiRNAのうち14.6%が有効と判定された(Fig. 1-2).ただし,この割合は遺伝子の種類によって変動し,Figure 1-5の赤色のヒストグラムに示すような広い分布を示した.たとえば,src遺伝子(SRC; NM_005417)では9.3%,EGF遺伝子(EGF; NM_001963)では17.9%のsiRNAが有効と判定された(Fig. 1-5).また,RefSeq release 16に登録されたヒトの全遺伝子に対してsiRNAの設計を試みたところ,すべての遺伝子に対して,すくなくとも1個の有効なsiRNAが設計可能であった.
Figure 1-5. ヒトの全遺伝子について,Ui-Teiらの設計法を満たすsiRNAの割合を求め,その分布を示した(赤色のヒストグラム).また,Ui-Teiらの設計法を満たすsiRNAのうち,Pol IIIプロモーターから発現させるshRNAとしても使用可能な配列の割合を求め,その分布を示した(青色のヒストグラム).ヒストグラムの階級の幅は0.3%である.src遺伝子およびEGF遺伝子における割合を例示した.
さらに,Pol IIIプロモーターをもつベクターからshRNAを発現させる場合,
-
センス鎖の5′末端がGであること.(このGから転写が開始されるため.)
-
配列中にUUUUもしくはその相補的配列であるAAAAを含まないこと.(途中で転写が終結することを防ぐため.)
が必要である(Ui-Tei et al., 2006).この条件を加えると,Ui-Teiらの設計法で有効と判定されるsiRNAは,ヒトの全遺伝子に対して設計可能なすべてのsiRNAのうちの5.7%となった.このとき,個々の遺伝子における割合はFigure 1-5の青色のヒストグラムのような分布となった.RefSeq release 16に登録されたヒトの全遺伝子に対してsiRNAの設計を試みたところ,99.9%以上の遺伝子に対して,すくなくとも1個の有効なsiRNAが設計可能であった.
以上の結果は,Ui-TeiらのsiRNA設計法が,ヒトの全遺伝子をほぼ網羅できるゲノムワイドなsiRNA設計に適用可能であることを示唆した.
1-3. siRNAの標的切断活性を評価するアッセイ系の構築
siRNAが本来の標的とは別の遺伝子の相同部分を認識し,その遺伝子を抑制してしまう現象は,siRNAのオフターゲット効果と呼ばれる(Jackson et al., 2004).本研究では,siRNA配列とmRNA配列との間にどの程度の相同性があるとオフターゲット効果が生じるかを検討するため,任意の標的配列に対してsiRNAの標的切断活性を評価できるアッセイ系(以下,標的切断アッセイ)を構築した.
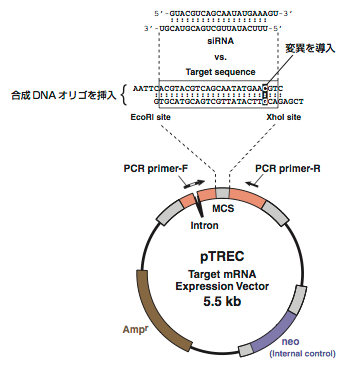
標的切断アッセイの概略は,siRNAの標的となる21〜40塩基の配列を組み込んだ発現ベクターとsiRNAとを同時に培養細胞に導入して,ベクターから発現する標的mRNAがsiRNAによって切断される効率を,リアルタイムRT-PCRで定量するというものである.本アッセイのために構築した標的発現ベクターpTREC(Target RNA Expression Constructの略)の概要をFigure 1-6に示す.本アッセイでは,合成DNAオリゴをpTRECベクターのMCSに挿入することによって標的配列を自由に設定できるため,ある特定のsiRNAに対して標的mRNAの配列を1塩基ずつ変えていくような系統的な解析が可能となる.たとえばFigure 1-6の例では,siRNAの標的部分に1塩基の置換を導入した場合の切断活性を評価できる.また,pTRECベクターから発現するmRNAの配列(Fig. 1-7)は標的部分を除き共通であるため,まったく同一の実験条件下でsiRNAの標的切断活性を定量的に評価できる.またRT-PCRをおこなう際も共通のPCRプライマーを使用するため,PCRの条件検討が不要でハイスループットなアッセイが可能である.
Figure 1-6. 本研究で構築した標的mRNA発現ベクターpTRECの構造.RT-PCRに用いるPCRプライマーの一方(PCR primer-F)の配列は,イントロンによって分断されるように設計した.この工夫により,PCRの際にプラスミドが鋳型となってDNAが増幅することを防ぐことができた.
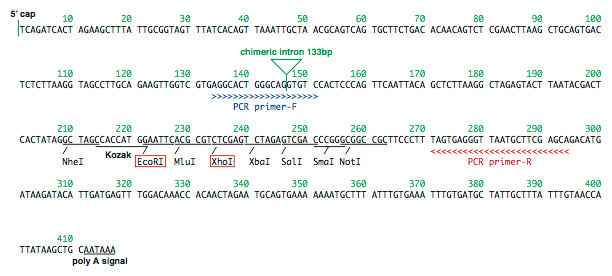
Figure 1-7. pTRECから標的mRNAとして転写される領域の塩基配列.本研究では,EcoRI部位とXhoI部位との間に合成DNAオリゴで作製した標的配列を挿入した.
まず本アッセイの有効性を検証するために,本アッセイの結果と,実際の標的遺伝子に対してsiRNAを用いた場合のRNAi活性とを比較した.
ホタルルシフェラーゼ遺伝子を標的とする9種のsiRNAについて本アッセイにより標的切断活性を評価し,一方でホタルルシフェラーゼ遺伝子に対するRNAi効果をルシフェラーゼアッセイで測定した.本アッセイで得られた結果は,実際にルシフェラーゼ遺伝子に対して各siRNAが示したRNAi活性と高い相関を示した(Fig. 1-8).両者の関係が完全に線形になっていない理由は,本アッセイがmRNAの量を評価しているのに対し,ルシフェラーゼアッセイは発現したタンパクの活性を評価しているという違いのためであろう.
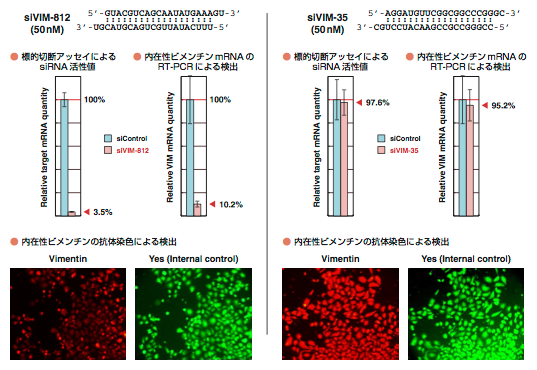
また,ヒト内在性遺伝子のビメンチン(VIM; NM_003380)を標的とする2種のsiRNAについて本アッセイにより標的切断活性を評価し,一方で内在性ビメンチンをノックダウンした効果をリアルタイムRT-PCRおよび抗体染色で評価した.本アッセイで得られた結果は,内在性ビメンチンに対して各siRNAが示したRNAi活性とよく一致した(Fig. 1-9).
以上の結果より,本アッセイがRNAi活性を評価する方法として有効であることが確認できた.
Figure 1-8. ホタルルシフェラーゼ遺伝子を標的とする9種のsiRNAを標的切断アッセイで評価した.標的切断アッセイの結果(縦軸)と,ホタルルシフェラーゼ遺伝子に対するRNAi効果(横軸)との間には高い相関が認められた.ルシフェラーゼ相対活性の値が低いほど,siRNAのRNAi活性が高いことを示している.
Figure 1-9. ヒト内在性遺伝子のビメンチンを標的とする2種のsiRNAを標的切断アッセイで評価した.標的切断アッセイの結果は,内在性ビメンチンをノックダウンしたときのmRNA定量値および抗体染色の結果と一致した.
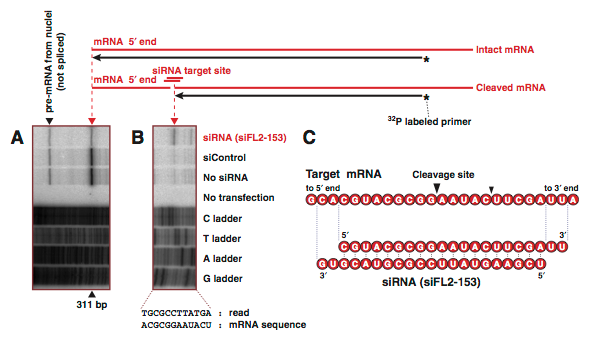
さらに,pTRECベクターから発現させた標的RNAのsiRNAによる切断を直接検出するために,プライマーエクステンション法による解析をおこなった.
ホタルルシフェラーゼ遺伝子に対するsiRNA(siFL2-153)と相同な23塩基を挿入した標的mRNAを,pTRECから発現させた.それと同時に,siFL2-153もしくは無関係な配列のsiRNA(siControl)を導入した場合について,poly A+ RNAを抽出してプライマーエクステンションをおこなった.その結果,いずれの場合も標的mRNAの5′末端に対応するバンドが予想された位置にあらわれた(Fig. 1-10A).また,RNA抽出の過程で核を分離していないため,イントロンを含むpre-mRNAの5′末端に対応するバンドも見られた.
標的mRNAにsiFL2-153を作用させた場合,核内のpre-mRNAのバンドに大きな変化はなかったが,mRNAの5′末端に対応するバンドが薄くなった(Fig. 1-10A).さらに,siRNAの標的配列部分に新たなバンドが生じた(Fig. 1-10B).このバンドは,siRNAによって切断されたmRNAのうち3′側のRNA断片の5′末端に対応するものと考えられる.すなわち,Figure 1-10Bのバンドの位置が,siRNAによる標的RNAの切断点であると考えられる.シークエンスラダーを参照すると,この切断点はsiRNAのアンチセンス鎖の5′末端から10番目の塩基と11番目の塩基の間の位置に対応することが判明した(Fig. 1-10C).この結果は,ショウジョウバエのin vitroでの実験における切断点と一致した(Elbashir et al., 2001b,c).なお,この切断点から3′側に5塩基離れた位置にも薄いバンドが存在し(Fig. 1-10B),この位置でも標的mRNAの切断が起こっている可能性が示唆された.この第2のバンドがアーティファクトである可能性を排除するため,異なる配列のsiRNAでも実験をおこなったところ,同様の位置に第2のバンドが確認された.また,siRNAのアンチセンス鎖の5′末端から7塩基をDNAに置換したキメラ型siRNAで同様の実験をおこなったところ,主バンドはそのままに,第2のバンドのみ消失した(データ示さず).
Figure 1-10. プライマーエクステンション法による標的mRNAの切断の検出.(A) mRNAおよびpre-mRNAの5′末端に対応するバンド.(B) siRNAによる切断点に対応するバンド.(C) 本実験のバンドの位置から求めた切断点を図示した.
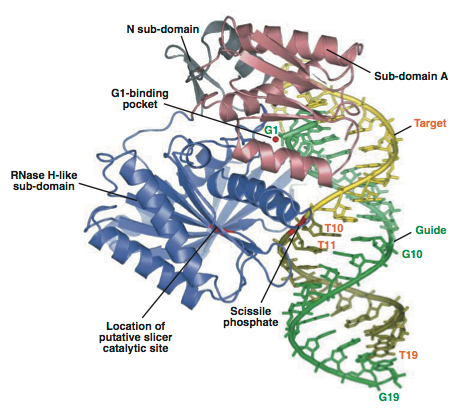
標的mRNAの切断点については,RISCを構成するタンパク質であるArgonauteがもつPIWIドメインの立体構造からも検討できる.ArgonauteのPIWIドメインはRNAiの実行過程において標的mRNAの切断活性を担っており(Yuan et al., 2005),RNaseHと類似した立体構造を有する(Song et al., 2004).Figure 1-11に示すように,Argonauteと結合したsiRNAのアンチセンス鎖(緑色)の5′末端(G1)はPIWIのポケットに収まり,siRNAのアンチセンス鎖と対合した標的mRNA(黄色)の切断点に相当する部分(赤色)が,RNaseHの活性中心とされているアミノ酸の近傍に位置している(Ma et al., 2005; Parker et al., 2005).しかしながら,この切断点から3′側に5塩基離れたヌクレオチドはPIWIドメインの反対側に位置しており,どのようなメカニズムでこの部分の切断が起こっているのか説明できず疑問が残った.
Figure 1-11. 硫酸還元古細菌Archaeoglobus fulgidusのPIWIドメインと,19塩基対の2本鎖RNAの複合体の立体構造(Parker et al., 2005より作成).緑色のRNA鎖がsiRNAのアンチセンス鎖,黄色のRNA鎖が標的mRNAに相当する.
1-4. オフターゲット効果が少ないsiRNA設計法の実験的検討(1)
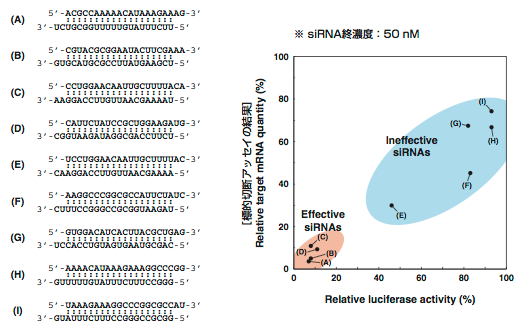
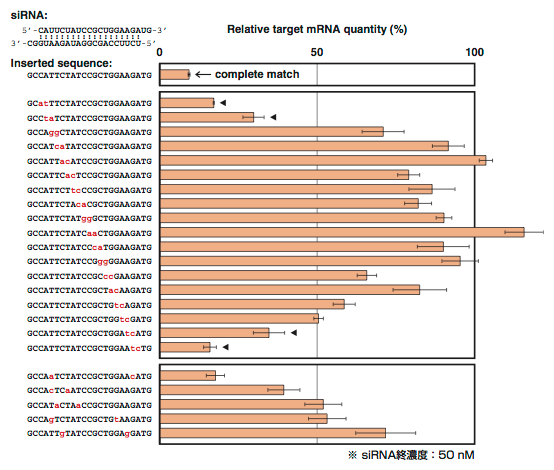
siRNA配列とmRNA配列との間にどの程度の相同性があるとオフターゲット効果が生じるかを明らかにするため,これまでの報告では,標的mRNAとは少しずつ配列が異なるsiRNAを合成しそれらのRNAi効果を評価する方法がとられてきた.しかしながら,化学合成のsiRNAは高価なこともあり,特定の遺伝子の限られた配列しか検討されていなかった.さらに,緒論でも述べたようにsiRNAの活性はその配列自体によって大きく変化するため,siRNAの配列を変えるという手段ではオフターゲット効果を独立に評価することはできない.オフターゲット効果を正確に評価するためには,siRNAの配列は変えずに,標的mRNAの配列を変化させる方法が必要である.そこで本研究では,ホタルルシフェラーゼ遺伝子を標的とするsiRNA(siFL-36)の配列に対して,様々なミスマッチを含む標的mRNAを発現するpTRECコンストラクトを系統的に構築した.
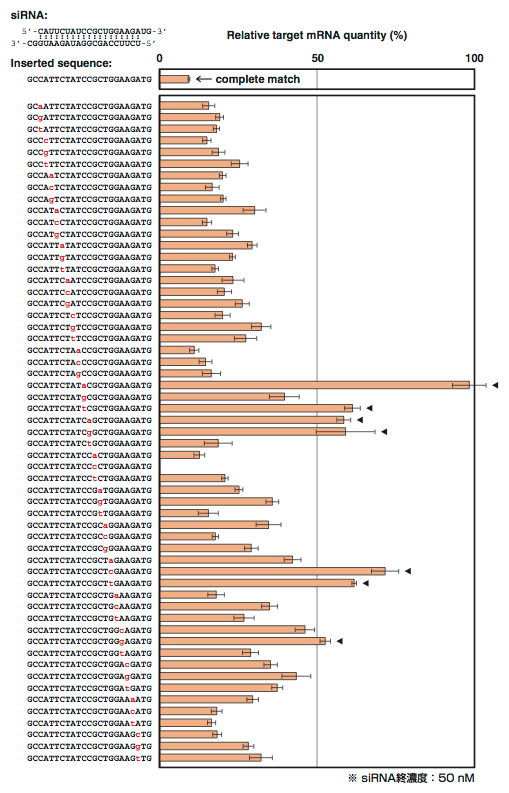
まず,siFL-36の標的配列に1塩基の置換を導入したすべての場合を検討した(Fig. 1-12).siRNAを50 nMで導入した場合,特に標的配列の中央付近の置換によりRNAi効果が失われた場合があったが(◀で表示),57例中49例について標的mRNAが50%以下まで分解された.siFL-36以外の配列でも検討をおこなったところ,やはり標的配列の中央付近の置換でRNAi効果が失われた場合があった(データ示さず).ただし,RNAi効果が失われるような置換の位置や,塩基の種類について規則性は見いだせなかった.標的配列の中央付近と末端付近とでミスマッチの効果が異なる事実は,他の報告(Du et al., 2005; Birmingham et al., 2006)でも指摘されている.
Figure 1-12. siFL-36の標的配列に1塩基の置換を導入した場合の,siFL-36によるRNAi効果.最上段は,siFL-36と相同な標的配列に対するRNAi効果(コントロール).siFL-36の配列および,pTRECに挿入した配列を図中に示した.
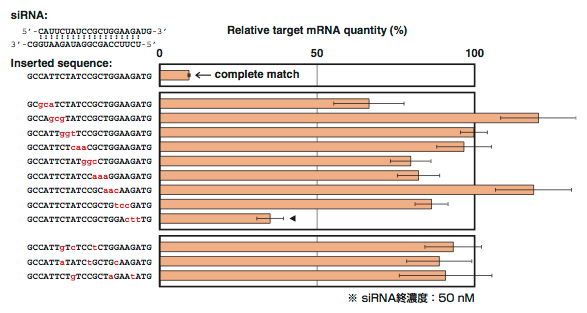
つぎに,siFL-36の標的配列に2塩基の置換を導入した23例を同様に検討した(Fig. 1-13).標的配列の中央付近に2塩基の連続する置換を導入した場合,RNAi効果がほぼ失われた.ただし,標的配列の末端付近の置換においては,標的mRNAが50%以下まで分解された場合があった(◀で表示).また,連続しない2塩基の置換を導入した場合,それらの位置や種類によってミスマッチの効果は異なっていた(Fig. 1-13).
さらに,siFL-36の標的配列に3塩基の置換を導入した12例を同様に検討した(Fig. 1-14).その結果,1例(◀で表示)を除くすべての例で,RNAi効果がほぼ失われた.
Figure 1-13. siFL-36の標的配列に2塩基の置換を導入した場合のRNAi効果.
Figure 1-14. siFL-36の標的配列に3塩基の置換を導入した場合のRNAi効果.
以上の結果を考慮すると,siRNA設計においては,標的と無関係なすべての配列に対してかならず3塩基以上がミスマッチする配列を選択することが望ましい.後述する情報科学的な解析からは,そのようなsiRNAは全体の約10%存在するが,4ミスマッチ以上を保証できるsiRNAはほとんど存在しない(Naito, Yamada et al., 2004).したがって,現実的なアプローチとして3ミスマッチ以上のsiRNAを選択することが妥当と考えられる.
なお,ミスマッチの位置によってその影響が異なる理由は,次のように考えられる.siRNAのアンチセンス鎖と標的mRNAは,A-formの2本鎖RNAを形成してPIWIドメインと結合している(Fig. 1-11).ところが,siRNAのアンチセンス鎖の中央部分が標的mRNAとミスマッチする場合,2本鎖の立体構造がみだれ,標的mRNAの切断点とPIWIドメイン活性中心との位置関係が変化すると予想される.その結果,標的mRNAは切断を受けなくなる可能性が高い.一方,ミスマッチの位置がsiRNAの末端部分の場合は,中央付近のミスマッチと比較して立体構造への影響は少なく,このため標的mRNAの切断活性も保持されるのであろう.
1-5. オフターゲット効果が少ないsiRNA設計法の実験的検討(2)
近年,siRNAを構成するアンチセンス鎖の5′末端から2〜8番目の塩基(以下,seedと呼ぶ)がmRNAと完全一致する場合に,翻訳の抑制およびmRNAの分解が起こりうることが示された(Lim et al., 2005; Lin et al., 2005; Birmingham et al., 2006).しかしながら,長さが7塩基のseedは計算上はmRNAの約16 kbに1箇所の割合で存在するため,seedの完全一致のみで遺伝子が抑制されるならば,膨大な数の遺伝子が影響を受けることになる.そこで本研究では,seedマッチによるオフターゲット効果を以下の方法で検討した.
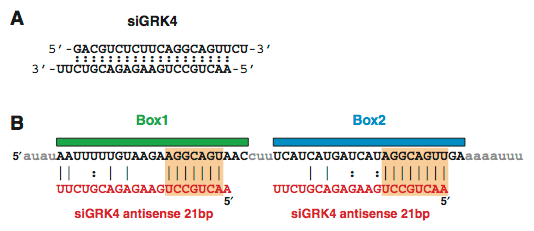
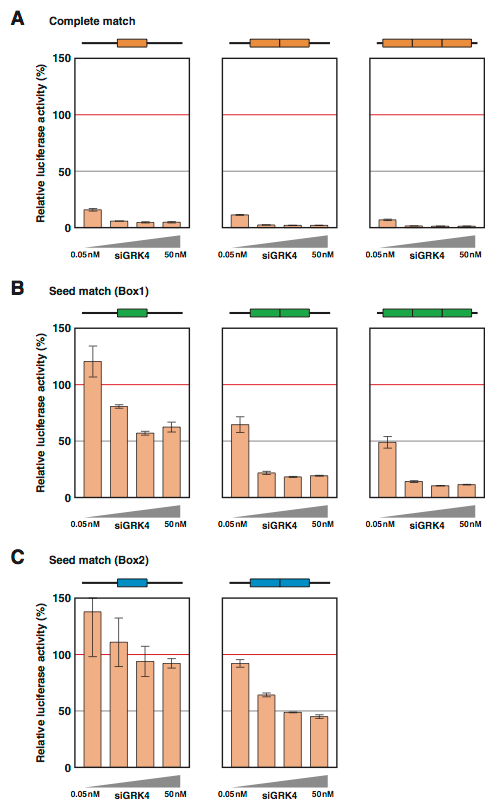
ヒトGRK4遺伝子に対するsiRNA(Fig. 1-15A; siGRK4)の配列と完全一致しないにも関わらず,siGRK4によって抑制される遺伝子として,Hif-1が報告されている(Lin et al., 2005).そこで,Hif-1遺伝子の3′ UTRから,siGRK4とseedマッチするBox1およびBox2(Fig. 1-15B)だけを取り出して,ウミシイタケルシフェラーゼ遺伝子の3′ UTRに挿入したコンストラクトを作製した.また同様に,Box1またはBox2をタンデムに2個もしくは3個挿入したコンストラクトを作製した.これらのコンストラクトと同時に,siGRK4を0.05〜50 nMの濃度でHeLa細胞に導入したときの,ウミシイタケルシフェラーゼ遺伝子の相対活性を測定した.
その結果,標的配列としてBox1を1個挿入した場合,siGRK4が5 nMまたは50 nMのときにルシフェラーゼ相対活性が50%近くまで抑制されたが,Box2を1個挿入した場合ではルシフェラーゼ相対活性の減少は見られなかった.一方,Box1を2個または3個挿入した場合,もしくはBox2を2個挿入した場合に,ルシフェラーゼ相対活性が強く抑制された(Fig. 1-16).
Figure 1-15. (A) siGRK4の配列.(B) ヒトHif-1遺伝子の3′ UTRに存在する,siGRK4とseedマッチする2つの配列.siGRK4のアンチセンス鎖とのアライメント中,seedマッチしている部分を影付きで示した.
Figure 1-16. (A) siGRK4と相補的な標的配列(黄色)に対してsiGRK4を作用させた場合の抑制効果.(B) Box1(緑色)を1つ,2つ,または3つ挿入した標的に対してsiGRK4を作用させた場合の抑制効果.(C) Box2(青色)を1つまたは2つ挿入した標的に対してsiGRK4を作用させた場合の抑制効果.いずれもsiGRK4は0.05, 0.5, 5, 50 nMの終濃度で導入した.
以上の結果は,わずか7塩基のseedマッチであっても,同一のmRNA分子内に複数のseedマッチが存在する場合に大きなオフターゲット効果が起こりうることを示唆した.したがってsiRNAの設計においては,標的とは無関係な転写産物のなかに複数のseedマッチが存在するようなsiRNA配列を避けることが望ましいと考えられた.ただし,RefSeq release 20に登録されたヒトの全遺伝子の配列を解析したところ,いかなる遺伝子においても同一の転写産物内に2回以上出現しないようなseedは,47種類のseedのうちわずか5種類しか存在しなかった.したがって,現実的にはより緩やかな基準を検討する必要がある.そのような方法のひとつとして,ヒトの転写産物において出現頻度の高いseedをなるべく避けるというアプローチが考えられる.
1-6. siRNA設計システムの構築
これまでに述べた実験的な結果に基づき,哺乳類細胞に有効かつオフターゲット効果の少ないsiRNAの設計システムを構築した.哺乳類細胞に有効なsiRNAの設計法は,Ui-Teiら,Reynoldsら,Amarzguiouiらの3つの設計法を比較検討した結果に基づき,Ui-Teiらの方法を用いた.また,オフターゲット効果の少ないsiRNAを選択するために全遺伝子に対する相同性検索をおこない,標的と無関係なすべての配列に対して3塩基以上がミスマッチする配列を可能なかぎり選択することとした.相同性検索には,当初BLAST(Altschul et al., 1990)を用いたが,BLASTはsiRNAのような短い配列の検索において見落としが多いという欠点があることが判った(Fig. 4).このため短い配列の検索を見落としなく高速に実行できるYamadaらのアルゴリズム(Yamada et al., 2005)を用いることとした.
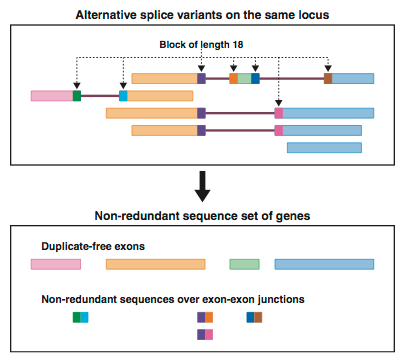
全遺伝子に対する相同性検索においては,RefSeqやGenBankなど既存のデータベースを用いると,同一の遺伝子に由来する配列が複数登録されているためsiRNAの特異性の評価が難しいという問題があった.たとえば,ヒトのsrc遺伝子はRefSeq release 20においてNM_005417およびNM_198291という2件の配列として登録されているため,srcを標的とするsiRNAの配列をRefSeq release 20に対して検索すると,この2件の配列がヒットしうる.このとき,2件のヒットが同一の遺伝子に由来するのか,それとも無関係な遺伝子を含むのかを機械的に判断することは困難である.そこで,非冗長な遺伝子配列データベース(NRDB; Non Redundant DataBase)に対してオフターゲット検索をおこなうこととした(Naito, Yamada et al., 2004).NRDBは,RefSeqおよびUnique UniGeneの配列をゲノムにマッピングして得られた非冗長なエクソン配列と,エクソンジャンクション配列とを統合した配列データベースである(Fig. 1-17).
Figure 1-17. NRDBの構築(Naito, Yamada et al., 2004より作成).冗長な配列をゲノムにマッピングすることによって,非冗長なエクソン配列およびエクソンジャンクション配列を得た.
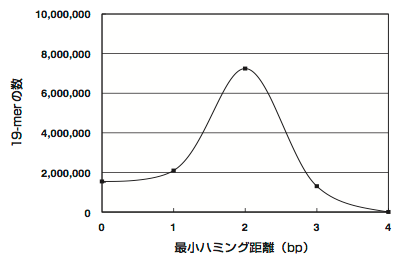
Yamadaおよび著者らは,ヒトのNRDBを構成する19-merの部分配列をすべて生成し,各配列のNRDBにおける最小ハミング距離を求めた(Naito, Yamada et al., 2004).最小ハミング距離とは,NRDB内でその配列を除きもっとも相同性の高い配列とのミスマッチ数である.たとえばある19-merの配列が複数の遺伝子に存在する場合,その19-merの最小ハミング距離は0である.また,ある19-merが他のすべての配列に対して必ず3塩基以上のミスマッチを有する場合,すなわち,その19-merと2ミスマッチ以内で相同な配列が他の遺伝子に存在しない場合,その19-merの最小ハミング距離は3である.なお,相補的(complementary)な配列も相同と見なす.
その結果,ヒトのNRDBを構成する19-merのうち最小ハミング距離が3以上である配列は10.7%であったが,4以上の配列はほとんど存在しなかった(Fig. 1-18).この結果は,siRNAを設計する際,標的と無関係なすべての配列に対して3塩基以上がミスマッチする配列は十分に選択可能であるが,4ミスマッチ以上を保証できる配列はほとんど選択できないことを示唆した.
Figure 1-18. ヒトのNRDBを構成する19-merの最小ハミング距離の分布(Naito, Yamada et al., 2004より作成).最小ハミング距離が5であるような19-merは存在しない.
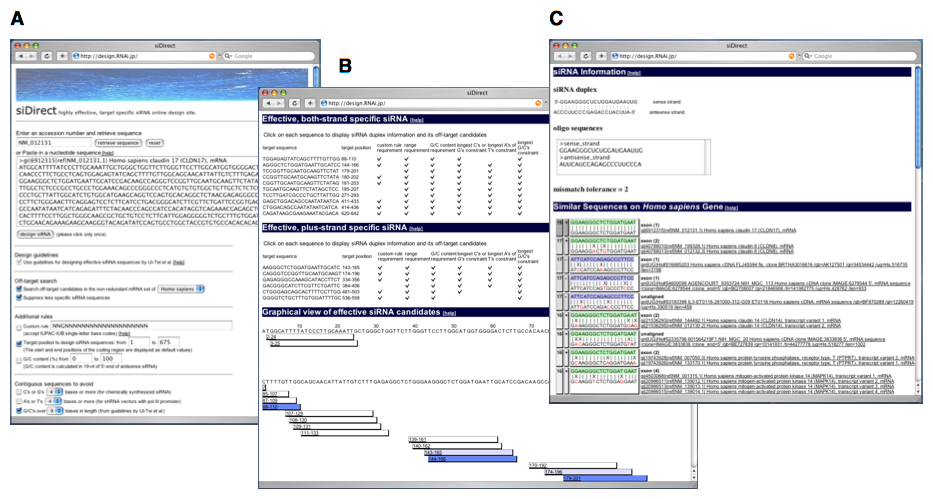
本研究で構築したsiRNA設計システムの一部は,siDirectウェブサーバ(Naito, Yamada et al., 2004; http://design.RNAi.jp/)として公開されており,その概略は下記のとおりである.
► トップページ(Fig. 1-19A)
基本的にはこの画面上に標的遺伝子の塩基配列またはアクセッション番号を入力するだけで,有効なsiRNAの配列が得られる.オフターゲット検索をおこなう生物種(ヒト,マウス等)を選択できるほか,各種のオプションが用意されている.
► siRNA設計結果の表示(Fig. 1-19B)
画面上のEffective, both-strand specific siRNAは,Ui-Teiらの設計法を満たし哺乳類細胞で有効と予測される配列で,かつsiRNAのセンス鎖,アンチセンス鎖とも,標的以外のすべての遺伝子に対して3塩基以上がミスマッチするようなsiRNA配列である.通常はこのリストからsiRNA配列を選択すればよい.これらのsiRNAは,画面上のGraphical view of effective siRNA candidatesでは濃い青色で表示される.
このようなsiRNAがひとつも設計できない場合はEffective, plus-strand specific siRNAを参照する.これらのsiRNAは,少なくともsiRNAのアンチセンス鎖については,標的以外のすべての遺伝子に対して3塩基以上がミスマッチするような配列である.この場合,siRNAのセンス鎖側の特異性は保証できないが,もしセンス鎖とハイブリダイズしうるオフターゲット遺伝子が存在しても,Ui-Teiらの設計法を満たすsiRNAのセンス鎖側は,RISCに効率よく取り込まれないと考えられるため(Ui-Tei et al., 2004),その遺伝子に対するオフターゲット効果はおそらく問題にならない.これらのsiRNAは,Graphical view of effective siRNA candidatesで薄い青色で表示される.
► オフターゲット遺伝子の表示(Fig. 1-19C)
Figure 1-19Bのリストで個々のsiRNA配列をクリックすると,そのsiRNAと3ミスマッチ以内で相同なすべての遺伝子が表示され,設計したsiRNAの特異性を確認できる(Fig. 1-19C).siRNAのアンチセンス鎖(緑)またはセンス鎖(青)と相同な部分がアライメントとして表示される.
Figure 1-19. siDirectウェブサーバ(http://design.RNAi.jp/).(A) トップページ.(B) ヒトのclaudin 17遺伝子(NM_012131)を標的とするsiRNAを設計した結果.(C) siRNA配列と3ミスマッチ以内で相同なヒト遺伝子の一覧.最上段には本来の標的遺伝子であるclaudin 17が19/19マッチで表示されている.またその下には,17/19マッチで本来の標的遺伝子とは無関係なclaudin 8が表示されている.
1-7. ヒト全遺伝子に対するsiRNA設計
本研究で構築したsiRNA設計システムにより,RefSeq release 20に登録されたヒトの全遺伝子に対してsiRNAの設計を試みた.各遺伝子について1個ずつ,下記の条件を可能なかぎり満たすsiRNAの配列を,配列表(附録)に示した.ただし,必須と表示されている条件は,かならず満たすこととした.
-
Ui-TeiらのsiRNA設計法を満たす,哺乳類で有効と予測される配列(必須).
-
Pol IIIプロモーターからshRNAを発現させる場合にも使用可能な配列(必須).
-
ヒトの当該遺伝子と対応する,マウスの遺伝子にも共通な配列.なお,共通な配列とは,siRNAのアンチセンス鎖部分の21塩基が完全一致していることを意味する.
-
選択的スプライシングによるバリアントが知られている場合,できるだけ多くのバリアントに共通な配列.
-
標的と無関係なすべての配列に対して3塩基以上がミスマッチする配列.
-
全遺伝子における出現頻度が低い7塩基をseedとする配列.
なお,配列表(附録)に記載した項目は下記のとおりである.
-
spe : Hs(ヒト)またはMm(マウス).
-
Gene ID : Entrez GeneID.
-
symbol : 遺伝子名.
-
accession : アクセッション番号.
-
position : siRNA配列の最初の塩基の位置.
-
target (21) + overhang (2) : siRNAのオーバーハングを含む23塩基の配列を,a, t, g, cの4文字で記載した.GとCの区別を容易にするため,すべて小文字で記載した.詳細はFigure 1-20を参照.
Figure 1-20. 配列表(附録)に記載の塩基配列と,実際のsiRNA配列との対応.
1-8. 長い2本鎖RNAから生成されるsiRNAのオフターゲット効果
化学合成したsiRNAを用いてRNAiを誘導する方法以外にも,長いdsRNAをDicerで処理して得た,さまざまな配列のsiRNAの混合物によってRNAiを誘導する方法がある(Kittler et al., 2005).また,チャイニーズハムスターのCHO-K1細胞では,長いdsRNAを直接用いてRNAiを誘導できるほか(Ui-Tei et al., 2000),ショウジョウバエや線虫など哺乳類以外の種では長いdsRNAが一般的に用いられている.長いdsRNAは細胞内でDicerによって約21塩基対のsiRNAに分解され,さまざまな配列のsiRNA混合物として標的mRNAに作用することとなる.このような場合,各siRNAと相同性の高い配列をもつ遺伝子は,すべてオフターゲット効果を受ける可能性がある.しかしながら,長いdsRNAによるオフターゲット効果を予測する方法としては,BLASTで単純にdsRNAの全長を検索する方法が一般的であった.
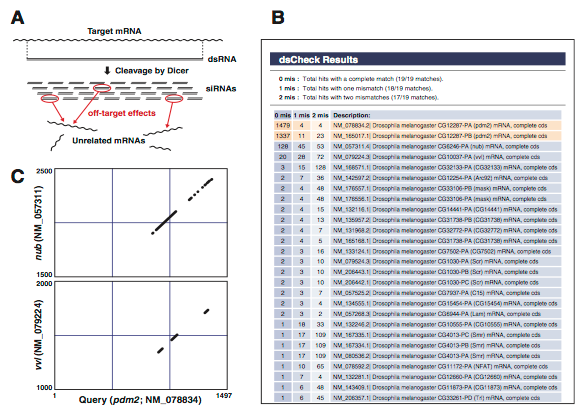
本研究では,長いdsRNAによるオフターゲット効果をより正確に予測するため,細胞内におけるRNAiの実行過程を反映したアルゴリズムを構築した.まず,細胞内でdsRNAがDicerによってsiRNAに分解されることを考慮して,dsRNA配列を構成する19塩基の部分配列(以下,単に19-merと呼ぶ)をすべて生成した(Fig. 1-21A).たとえば,500塩基の長さのdsRNA配列からは,482種の19-merが生成できる.このようにして仮想的に生成した各siRNAについて個別にオフターゲット検索をおこない,完全マッチ(19/19マッチ),1ミスマッチ(18/19マッチ),2ミスマッチ(17/19マッチ)で相同なすべての遺伝子を列挙して集計した(Fig. 1-21B).実際にはdsRNA配列を構成するすべての部分配列がsiRNAとして生成されるわけではないが,オフターゲット効果の対象となりうる遺伝子を見落としなく列挙するため,本研究ではすべての部分配列が生成されると仮定して解析をおこなった.なお,部分配列の長さを21塩基ではなく19塩基とした理由は,siRNAの末端の塩基が標的mRNAとミスマッチしていても,中央の19塩基が完全一致していれば標的の切断が起こりうるためである.
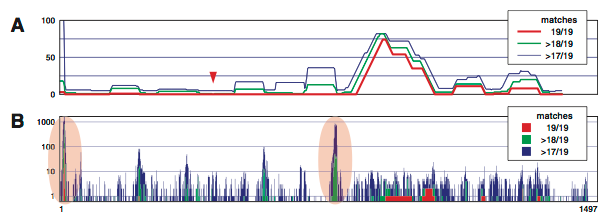
Figure 1-21Bは,ショウジョウバエのPOUドメインタンパクであるpdm2遺伝子(NM_078834)のコード領域の全長(1497 bp)を標的とするdsRNAについて,この方法でオフターゲット効果を予測した結果である.最上段から順に,pdm2遺伝子の2つのバリアント(NM_078834, NM_165017)および,無関係な遺伝子である,nub遺伝子(NM_057311),vvl遺伝子(NM_079224)がヒットしている.Figure 1-21Cに示すように,pdm2, nub, vvlの各遺伝子は,高度に保存されたPOUドメインを有しており,この領域を標的とするdsRNAは,nub遺伝子やvvl遺伝子に対するオフターゲット効果のリスクが高いと予想される.
Figure 1-21. Naito, Yamada et al., 2005より作成.(A) 長いdsRNAは細胞内でDicerによって約21塩基対のsiRNAに分解される.(B) ショウジョウバエpdm2遺伝子の全長を標的とするdsRNAのオフターゲット効果を予測した例.最上段には本来の標的遺伝子であるpdm2遺伝子の2つのバリアントが表示されている.その下にはオフターゲット遺伝子の候補として,nub遺伝子,vvl遺伝子などが表示されている.(C) pdm2遺伝子,nub遺伝子,vvl遺伝子は,保存されたPOUドメインを共有している.17/19以上のマッチをプロットした.
このような遺伝子に対して特異性が高いdsRNAを設計するためには,特定のオフターゲット遺伝子に対して多数の19-merがヒットする協調的ヒット(collaborative off-target hit)を避けることが望ましい(Naito, Yamada et al., 2005).Figure 1-22Aは,pdm2遺伝子に対して100 bpのdsRNAをさまざまな位置に設計した場合に,dsRNAから生成された82種の19-merによる協調的ヒットの最大値を計算したものである.POUドメインが存在する1000 bp付近にピークがあり,この付近ではdsRNAから生成した19-merの多くが特定のオフターゲット遺伝子(この場合はnub遺伝子)と相同性を有することを示している.一方,500 bp付近の矢印(▼)で示した位置は協調的ヒットが少なく,100 bpのdsRNAを設計するうえで好適と考えられる.またFigure 1-22Bの影付で示すように,特定の19-merが多数の遺伝子と相同性を有する場合があり,そのような19-merを含まないようにdsRNAを設計することが望ましいと考えられる.
本研究で構築した,長いdsRNAによるオフターゲット効果の予測法と,特異性が高いdsRNAの設計法の一部は,dsCheckウェブサーバ(http://dsCheck.RNAi.jp/)として公開されている(Naito, Yamada et al., 2005).
Figure 1-22. pdm2遺伝子に対して特異性の高いdsRNAを設計する方法の検討(Naito, Yamada et al., 2005より作成).(A) 100 bpのdsRNAから生成した82種の19-merによる,協調的ヒットの最大値.矢印で示した位置が最も協調的ヒットが少なくなる.(B) 各19-merと相同性をもつ遺伝子の数.影付で示した位置の19-merは,多数の遺伝子と相同性を有する.
2. 方法
2-1. Ui-TeiらによるsiRNA設計法
Ui-Tei et al., 2004にしたがい,下記(i)〜(iv)の条件をすべて満たすsiRNAを有効と判定した.Figure 1-1Aに(i)〜(iv)を図示した.以下,siRNAを構成するセンス鎖,アンチセンス鎖を,それぞれ単にセンス鎖,アンチセンス鎖と呼ぶ.
(i) アンチセンス鎖の5′末端がAまたはUである.
(ii) センス鎖の5′末端がGまたはCである.
(iii) アンチセンス鎖の5′端の7塩基のうち4塩基以上がAまたはUである.
(iv) 配列中にG/Cが10塩基以上連続しない.
2-2. ReynoldsらによるsiRNA設計法
Reynolds et al., 2004にしたがい,下記(I)〜(VIII)の合計得点が6以上のsiRNAを有効と判定した.Figure 1-1Bに(I)〜(VIII)を図示した.
(I) siRNAの2本鎖部分におけるG/Cの数が,19塩基中7〜10塩基の範囲であれば+1点.
(II) アンチセンス鎖の5′端の5塩基におけるAまたはUの数(0〜5)を得点とする.
(III) 2本鎖部分のTmが20℃未満.判定不能のため本項目は無視する.
(IV) センス鎖の5′端から19番目の塩基がAであれば+1点.すなわち,アンチセンス鎖の5′末端がUであれば+1点.
(V) センス鎖の5′端から3番目の塩基がAであれば+1点.
(VI) センス鎖の5′端から10番目の塩基がUであれば+1点.
(VII) アンチセンス鎖の5′末端がGまたはCであれば-1点.
(VIII) センス鎖の5′端から13番目の塩基がGであれば-1点.
2-3. AmarzguiouiらによるsiRNA設計法
Amarzguioui et al., 2004にしたがい,下記(1)〜(6)の合計得点が3以上のsiRNAを有効と判定した.Figure 1-1Cに(1)〜(6)を図示した.
(1) ΔT3:アンチセンス鎖の5′端の3塩基におけるAまたはUの数(0〜3)から,センス鎖の5′端の3塩基におけるAまたはUの数(0〜3)を引いた値を得点とする.-3から+3までの値を取りうる.
(2) センス鎖の5′末端がGまたはCであれば+1点.
(3) センス鎖の5′末端がUであれば-1点.
(4) センス鎖の5′端から6番目の塩基がAであれば+1点.
(5) センス鎖の5′端から19番目の塩基がGであれば-1点.すなわち,アンチセンス鎖の5′末端がCであれば-1点.
(6) アンチセンス鎖の5′末端がAまたはUであれば+1点.
2-4. siRNA
Proligo社より購入したセンス1本鎖RNAとアンチセンス1本鎖RNAをアニーリングして使用した.アニーリングは,終濃度10 mM Tris-HCl (pH 7.5),20 mM NaClの緩衝液中で95℃で3分インキュベートし,徐々に室温まで冷却した.もしくは,2本鎖にアニーリングされたsiRNAをRNAi社(東京)より購入した.siRNAの配列はTable S1(巻末),Figure 1-8, 1-9, 1-10C, 1-12, 1-13, 1-14, 1-15Aにそれぞれ示した.
2-5. 細胞の培養およびトランスフェクション
HeLa細胞は10%非働化ウシ胎児血清(三菱化学)を含有するDulbeccoユs modified Eagle medium(DMEM; Gibco社)に抗生物質(10 U/mlペニシリン,50 g/mlストレプトマイシン)を添加して37℃で培養した.HeLa細胞へのトランスフェクションは,24ウェルプレートにおいて,Lipofectamine 2000(Invitrogen社)を用いて同製品のマニュアルに記載の方法でおこなった.細胞に導入するプラスミドは,Qiagen Plasmid MidiまたはQIAwell 96 Ultra(Qiagen社)を用いて抽出した.
2-6. ルシフェラーゼ遺伝子を標的とするRNAi
ホタルルシフェラーゼ遺伝子をコードするpGL3-Control(Promega社)を1 g/well,内部標準としてウミシイタケルシフェラーゼ遺伝子をコードするpRL-TK(Promega社)を0.1 g/well,siRNAを終濃度50 nMでHeLa細胞に導入した.24時間後に細胞を回収し,Dual-luciferase reporter assay system(Promega社)でルシフェラーゼ相対活性を測定した.
2-7. 標的mRNA発現ベクターpTRECの構築
pCI-neo(Promega社)のNheI部位とXbaI部位との間に,Kozak配列およびいくつかの制限酵素部位を挿入してpTRECを構築した.挿入した配列は下記のとおりである.
[NheI site]-CACCATGGAATTCACGCGTCTCGAG-[XbaI site]
構築したベクターの構造はFigure 1-6に,標的mRNAとして転写される領域の塩基配列はFigure 1-7に示した.
2-8. 標的切断アッセイ
pTRECベクターのEcoRI部位とXhoI部位との間に,評価対象のsiRNAと相同な23塩基の標的配列をFigure 1-6の例にしたがって挿入した.ただし,siRNAの配列に対してミスマッチを含む標的配列は,Figure 1-12, 1-13, 1-14に示した配列をそれぞれ挿入した.ベクターに挿入した標的配列部分はすべてシークエンスの確認をおこなった.
構築したpTRECコンストラクト(0.5 g/well)をsiRNA(終濃度50 nM)とともにHeLa細胞に導入し,24時間後に細胞を回収後,RNeasy96(Qiagen社)で全RNAを抽出した.得られたRNAを,オリゴdTをプライマーにSuperScript(Invitrogen社)で逆転写してcDNAを得た.リアルタイムPCRは,ABI PRISM 7000(Applied Biosystems社)を使用してSYBR Green PCR Master Mix(Applied Biosystems社)でおこなった.プライマーの配列は,
Target-F : 5′-AGGCACTGGGCAGGTGTC
Target-R : 5′-TGCTCGAAGCATTAACCCTCACTA
neo-F : 5′-ATCAGGATGATCTGGACGAAG
neo-R : 5′-CTCTTCAGCAATATCACGGGT
である.ベクターに組み込まれているネオマイシン耐性遺伝子を内部標準として使用し,[標的mRNA量] / [ neo mRNA量]により算出される値を,標的mRNA相対量とした.
2-9. ビメンチン遺伝子を標的とするRNAi
抗体染色によるビメンチンタンパク質の検出においては,HeLa細胞を24ウェルプレートにまき,1日後,2日後,3日後にそれぞれsiRNAを終濃度50 nMで導入した.3回目の導入操作から24時間後に,細胞を3.7%ホルムアルデヒドで固定し,PBSで洗浄した.ビメンチンの検出は,抗ブタビメンチン抗体(Oncogene Research Products社)およびCy3標識2次抗体(Jackson Immuno Research社)を用いた.またコントロールとしてYesタンパク質(YES1; NM_005433)の検出を,抗ヒトYes抗体(Upstate Biotechnology社)およびCy5標識2次抗体(Jackson Immuno Research社)でおこなった.
リアルタイムRT-PCRによるビメンチンmRNAの定量においては,HeLa細胞にpCAGIPuro-EGFP(ピューロマイシン耐性遺伝子およびEGFPをコードするベクター)を0.5 g,siRNAを終濃度50 nMで導入した.24時間後より培地をピューロマイシン含有培地に変更して細胞を培養した.導入3日後に標的切断アッセイと同様の方法でHeLa細胞からRNAを抽出し,ビメンチンのmRNA量および,内部標準としてβ−アクチン(ACTB; NM_001101)のmRNA量を定量した.ビメンチンおよびβ−アクチンの検出に用いたプライマーの配列はそれぞれ,
VIM-F : 5′-GAGCTACGTGACTACGTCCA
VIM-R : 5′-GTTCTTGAACTCGGTGTTGAT
ACTB-F : 5′-CACACTGTGCCCATCTACGA
ACTB-R : 5′-GCCATCTCTTGCTCGAAGTC
である.[ VIM mRNA量] / [ ACTB mRNA量]の値をビメンチンmRNA相対量とした.
2-10. プライマーエクステンション法
pTRECコンストラクトを0.5 g/well,siRNAを終濃度5 nMでHeLa細胞に導入した.24時間後,1サンプルあたり48ウェルから細胞を回収し,QuickPrep Micro mRNA Purification Kit(Amersham社)でpoly A+ RNAを抽出した.抽出したRNAをテンプレートに,32Pで末端標識したプライマー5′-CTCGAAGCATTAACCCTCACTAを用いて,Primer Extension System(Promega社)でプライマーエクステンション反応をおこなった.反応産物はシークエンスゲル(尿素−6%ポリアクリルアミドゲル)で泳動した.マーカーとして,同じプライマーを用いてpTRECコンストラクトをダイデオキシ法によりシークエンスして得たラダーを泳動した.
2-11. seedマッチによるオフターゲット効果の検討
psiCheck-1ベクター(Promega社)のXhoI部位とEcoRI部位との間に,Table 1-1の配列を挿入したコンストラクトを構築した.構築したコンストラクトを0.1 g/well,pGL3-Control(Promega社)を1 g/well,siRNAを終濃度0.05〜50 nMでHeLa細胞に導入した.24時間後に細胞を回収し,Dual-luciferase reporter assay system(Promega社)でルシフェラーゼ相対活性を測定した.
Table 1-1. seedマッチによるオフターゲット効果の検討のため,psiCheck-1ベクターに挿入した配列の一覧.
2-12. 共同研究に関する事項
Table S1(巻末)のうちsiRNAの名称に*を表示した箇所はUi-Tei et al., 2004によった.また,それ以外の約半数のデータを東京大学の程 久美子博士より供与いただいた.それ以外の箇所は著者が実験をおこない,全体にわたって著者が主体となって分析および検証をおこなった.
Figure 1-8のルシフェラーゼ相対活性のデータは,東京大学の程 久美子博士,江田 理愛氏より供与いただいた.それに対応する標的切断アッセイは著者がおこない,全体にわたり著者が主体となって分析および検証をおこなった.
Figure 1-9の内在性ビメンチンタンパク質の抗体染色のデータは,東京大学の高橋 史峰博士より供与いただいた.それに対応する標的切断アッセイおよび内在性ビメンチンmRNAの定量は著者がおこない,全体にわたり著者が主体となって分析および検証をおこなった.
NRDBの構築およびsiDirectウェブサーバのプログラム作成は東京大学の山田 智之氏,森下 真一博士がおこなった.NRDBの概念およびsiDirectウェブサーバの仕様は著者が主体となって発案,構成したほか,siDirectウェブサーバのプロトタイプを著者が作成した.また,構築時における評価およびデバッグを著者が主体となっておこなった.
dsCheckウェブサーバのプログラム作成は東京大学の山田 智之氏,松宮 孝大氏,森下 真一博士がおこなった.dsCheckウェブサーバの仕様は著者が主体となって発案,構成したほか,dsCheckウェブサーバのプロトタイプを著者が作成した.また,構築時における評価およびデバッグを著者が主体となっておこなった.
第二章 抗ウイルスsiRNA設計システムの構築およびHIV-1を標的とするRNA干渉法への応用
Trackbacks:0
- Trackback URL for this entry
- /phd/p2/trackback
- Listed below are links to weblogs that reference
- 第一章 from mesoの実験ノート