Home > Archives > 2011-07
2011-07
Nを含む塩基配列の検索、DB追加+アップデート
- 2011-07-29 (金)
- DBCLS
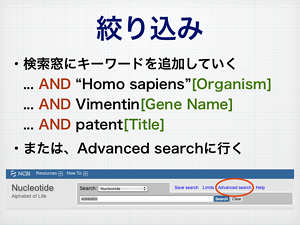
Nを含む塩基配列の検索
GGRNAで塩基配列を検索する場合、今まで seq1: や seq2: のタグでそれぞれ1塩基または2塩基以内のミスマッチを許容する検索ができましたが、AGGTCANNNTGACCT のようにあいまいな塩基を含む検索には対応していませんでした。今回のアップデートでは、iub:AGGTCANNNTGACCT(→GGRNAで検索)とすることで、あいまいな塩基(IUBコード)を含む配列を検索できるようにしました。狙いは、RNA結合タンパクが認識するあいまいなモチーフを検索したり(例:PUM結合サイト iub:UGUANAUA →GGRNAで検索;10秒くらいかかる)、GGRNAのエンジンをゲノム検索に応用して転写因子結合サイトなどを探すことを想定しています。(そのためには、もっとタンパクが核酸とくっつく気持ちで配列検索できるようなプログラムを書きたい・・・)
(参考:IUBコード表)
| Code | Base | Comment |
|---|---|---|
| R | A, G | puRines |
| Y | C, T | pYrimidines |
| M | A, C | aMino |
| K | G, T | Keto |
| S | C, G | Strong |
| W | A, T | Weak |
| H | A, C, T | not G |
| B | C, G, T | not A |
| V | A, C, G | not T |
| D | A, G, T | not C |
| N | A, C, G, T | aNy |
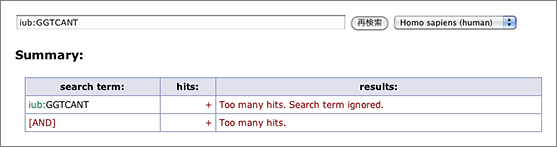
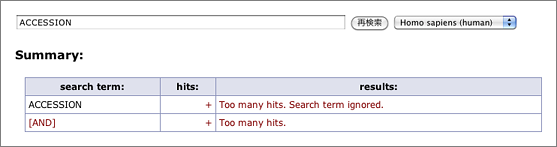
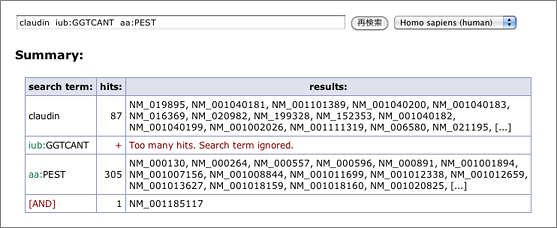
N等のあいまいな塩基が多くなるほどヒット件数が爆発的に増えて検索に時間がかかります。そこで今回のアップデートでは、一つのキーワードがヒットするRefSeq IDの件数が10,000を越える場合、そのキーワードは「絞り込みに寄与しない」と見なして無視するようにしました。具体的に説明すると、たとえば iub:GGTCANT では15,000件以上のRefSeq IDがヒットします。また ACCESSION で検索すると、(RefSeqのデータには必ずACCESSIONという文字列が含まれているので)今までは全件がヒットしていました(泣)。今後は、このようなキーワードを単独で検索すると “Too many hits.” として何も表示せず、またAND検索ではそのキーワードを含めずに検索した結果を返すことにしました。
(iub:GGTCANT で検索 → “Too many hits.”)
(ACCESSION で検索 → “Too many hits.”)
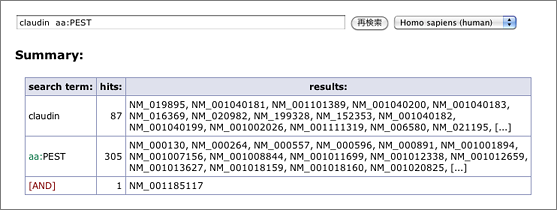
(claudin iub:GGTCANT aa:PEST で検索 → claudin aa:PEST で検索した場合と同じ結果)
ツメガエル、イネの追加
Twitterなどで要望をいただいたツメガエルXenopus (Silurana) tropicalisとイネOrysa sativaを追加。ただし中身を眺めてみると、ツメガエルはRefSeq IDがXM_,XR_で始まる “PREDICTED: ~” な遺伝子のほうが多いし(23,899件中15,110件)、イネは遺伝子名がついておらずsymbolがすべてOs02g0617400のような名前になっています。今後のRefSeqのアップデートに期待したいです。
RefSeqアップデート
Release 48 (Jul, 2011) にアップデートしました。RefSeqは2ヶ月に1回のペースでリリースされるので、更新の作業もだいぶ自動化しました。余談ですが、データをダウンロードするのにlftpのpgetを利用すると、1つのファイルを分割して各パートを並列ダウンロードするのでかなり速くなります。
lftp -c "mirror -i rna.gbff --parallel=3 --use-pget-n=3 \
ftp://ftp.ncbi.nlm.nih.gov/refseq/release/complete RefSeq_DL/"
- 「mirror」はディレクトリをまるごとミラーするlftpのコマンド。
- 「-i rna.gbff」はファイル名に rna.gbff を含むものだけをダウンロード。
- 「–parallel=3」は3ファイルを並列ダウンロード。
- 「–use-pget-n=3」は1つのファイルを3分割して並列ダウンロード。
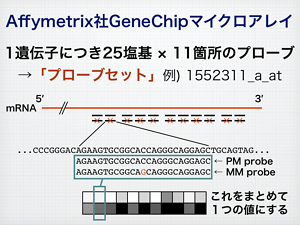
マイクロアレイのプローブ情報を追加
ツメガエルとイネのデータを追加したことに伴い、以下のアレイのプローブ情報を追加しました。
| メーカー | 生物種 | マイクロアレイの種類 | GPL ID |
|---|---|---|---|
| Affymetrix | ツメガエル | Xenopus laevis Genome Array | GPL1318 |
| Affymetrix | ツメガエル | Xenopus laevis Genome 2.0 Array | GPL10756 |
| Affymetrix | ツメガエル | Xenopus tropicalis Genome Array | |
| Affymetrix | イネ | Rice Genome Array | |
| Agilent | ツメガエル | X. laevis (Frog) Oligo Microarray v2 (023448) | |
| Agilent | ツメガエル | Xenopus laevis (Frog) Oligo Microarray (013665) | |
| Agilent | ツメガエル | Xenopus Gene Expression Microarray (015066) | GPL11258 |
| Agilent | イネ | O. sativa (Rice) Oligo Microarray 4x44K (015058) | |
| Agilent | イネ | Rice Gene Expression Microarray (015241) | GPL6864,GPL8852 |
| Agilent | イネ | Rice Microarray (012106) | GPL892,GPL7268 |
今後の方針もろもろ
昨日、@h_ono さんの出身ラボの方々からGGRNAについてフィードバックを頂きました。インターフェースで改善すべき点からGGRNA自体の位置付けまで、ウェットな現場からの辛口な、しかしどれも納得のご指摘でした。やはり現場で「使える」ものでなければ作る意味がないわけで、あくまで生物学を解くことが開発の目的であるということを忘れないようにしたい。それを踏まえ、GGRNAが生命科学研究にどう使えるのかを、NCBIのサイトではできないことを中心に次回紹介したいと思います。
- Comments (Close): 0
- Trackbacks: 0
統合データベース講習会 AJACS本郷9
- 2011-07-19 (火)
- DBCLS
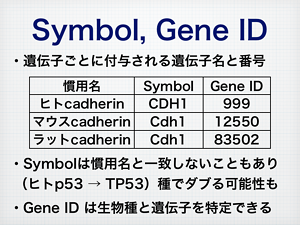
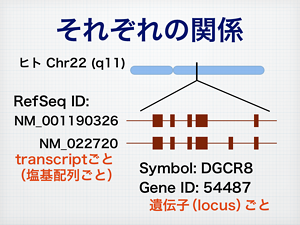
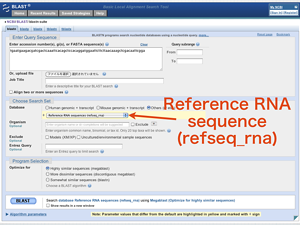
統合データベース講習会AJACS本郷9で「RefSeqの検索とRefEx(Reference Expression dataset)」という話をしました。「遺伝子をさがす」をテーマに、mesoが開発中のGGRNAと、センターで提供しているRefExの紹介をしました。
- AJACS本郷9 講習会ページ — http://motdb.dbcls.jp/?AJACS27
- 統合遺伝子検索GGRNA —
http://bit.ly/GGRNA→ http://GGRNA.dbcls.jp/ (8/18更新:URLを変更) - RefEx — http://togoexp.dbcls.jp/aboutrefex
(8/1 追記)統合TVに講習会の動画がアップされました!
- 統合TV:RefSeqの検索とRefEX — http://togotv.dbcls.jp/20110723.html
前職のときに一般参加者として出たこともあった講習会で、講師として話すことになるとは感慨深いです。デモに手間取ったりして肝心の後半部分が駆け足になってしまうなど不慣れな点もありましたが、話す機会をいただけたことに感謝します。
講習会で使用したスライドをここにアップしておきます。上記のAJACS本郷9 講習会ページとあわせて参照ください。
- mesoスライドPDF (3.2MB)
- Comments (Close): 0
- Trackbacks: 0
GGRNA英語版の公開&ヒット位置の表示
- 2011-07-05 (火)
- DBCLS
GGRNA英語版の公開
英語版のページもつくりました。英語がおかしなところを見つけたらご指摘いただければありがたいです。
http://altair.dbcls.jp/GGRNA/en/→ http://GGRNA.dbcls.jp/en/ (8/18更新:URLを変更)
そろそろヘルプのページも用意しないと。誰か統合TVで紹介してくれないかな(笑
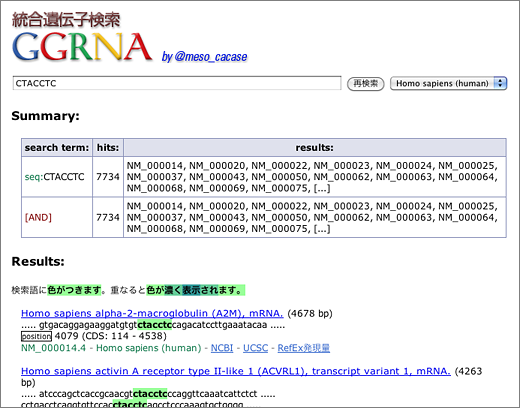
配列がヒットした位置を表示
GGRNAの売りは配列をさくさく検索できること。今回のアップデートでは、塩基配列またはアミノ酸配列がヒットした場合に位置を表示する機能をつけました。塩基配列の場合は参考としてCDSの範囲を表示することによって、ヒットした配列がCDS内かUTRかを簡単に判断できるようにしました。
応用:miRNAとseedマッチする遺伝子の探索
ヒットした位置を表示する機能はけっこう使えると思います(自画自賛)。ここでは応用例として、ヒトのmiRNAであるlet-7bの ‘seed’ と相補的な配列を3′ UTRにもつtranscriptのリストを作成してみます。このリストの一部にlet-7bの標的が含まれていると考えられます。
ヒトのlet-7b:5′-UGAGGUAGUAGGUUGUGUGGUU-3’(miRBaseより)
赤字で示した、5’末端から数えて2-8の位置のGAGGUAGがこのmiRNAのseedと呼ばれる部位です。miRNAはまずこの部分で相手を認識すると考えられており、miRNAの標的となるmRNAの多くはその3′ UTRに「seedと相補的な配列」をもっています。ここで留意しておきたいのは、
- seedの対合はperfect matchでなければならない。
RNA同士の塩基対形成なのですが、G:Uはダメです。 - 3′ UTRにseed matchするものすべてが標的となるわけではない。
実際に検索してみるとわかりますが、7塩基のexact matchだと数千個のtranscriptがヒットします。ですが、このすべてがmiRNAによって抑制されるわけではありません。ものすごくおおざっぱに言うと、マイルドに抑制されるものがこのうち数十個、きっちり抑制されるものは数個以下、というイメージではないかと思います。
それでは実際に検索してみましょう。GAGGUAGと塩基対形成するmRNAの配列はCUACCUC、ただしRefSeqのデータはmRNAといいながらも[ATGC]で表記されているので、CTACCTCを検索します。結果は7734件。ヒット件数が多いので結果が表示されるまで数秒かかります。(検索自体は2秒以内に終わるのですが、HTML生成とブラウザでの表示に時間がかかっているようです。ここは工夫できそう。)
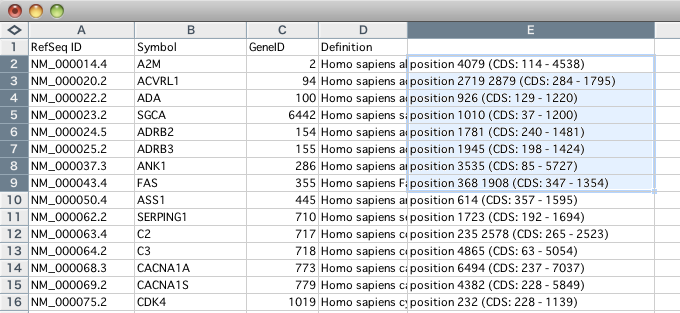
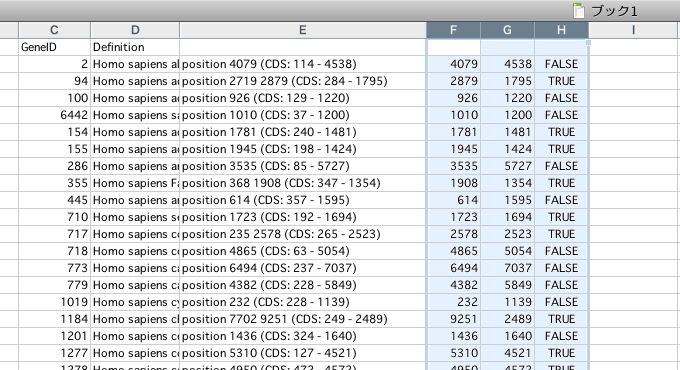
このなかには、今回探している3′ UTRにマッチしているもの以外に、5′ UTRやCDSにマッチしているものも含まれています。そこで今回の新機能、ヒット位置の情報をもとに3′ UTRにマッチしているものだけをエクセルで抜き出します。なお、grepとかawkとかperlとかRとかを使いこなせる人は、そっとエクセルを閉じてください。
検索結果のページの最下部にあるタブ区切りテキストをエクセルにコピペします。
E列のpositionの情報とCDSの位置を使います。3′ UTRにヒットしているもの、つまり(ヒット位置)>(CDSの終わりの位置)の行だけを取り出したいので、編集メニューの置換機能を利用してこの2つの数値を抜き出します。なお複数ヒットしているものは最後の(一番大きい)数値だけを判定すれば十分です。
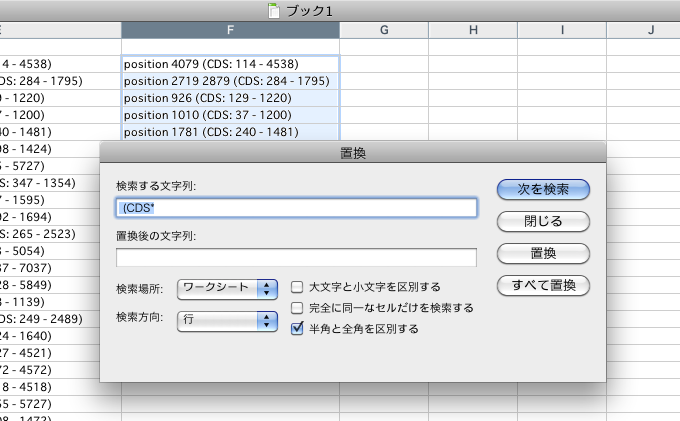
ヒット位置の最後の数値を取り出すには、position….のセルに対して、
- ” (CDS*” を空白で置換(1文字目のスペースに注意)
- “* ” を空白で置換(*の後のスペースに注意)
CDSの終わりの位置を取り出すには、同様にposition….のセルに対して、
- “)*” を空白で置換
- “* “を空白で置換(*の後のスペースに注意)
でOKですね。
取り出した2つの数値の大小をH列にて比較。
並べ替えを利用してTRUEとなっている行を抽出すると2414行。目的のリストが得られました。
なお、この作業でつかれてしまった人は、awkやperlで同じことが3行くらいで書けるのでそちらをおすすめします。
cat ggrna_result.txt | perl -ne '$_ =~ /(\d+) \(CDS: \d+ - (\d+)\)/ and $1 > $2 and print'
うそです1行で書けました。結果をwcするとエクセルで抽出した場合と同様に2414行になっています。
- Comments (Close): 0
- Trackbacks: 0
Home > Archives > 2011-07
- Search
- Feeds
- Meta