- 2014-08-01 (金) 15:46
哺乳類細胞で有効かつ標的遺伝子に特異的なsiRNA設計法の確立とその抗ウイルスRNA干渉法への応用
Antiviral RNA interference based on the development of the highly effective, target-specific siRNA design in mammalian cells
平成18年12月 博士(理学)申請
東京大学大学院理学系研究科 生物化学専攻 内藤 雄樹
・学位論文 (1) – 要旨・緒論
・学位論文 (2) – 第一章
・学位論文 (3) – 第二章(本記事)
・学位論文 (4) – 文献・巻末表・謝辞
第二章 抗ウイルスsiRNA設計システムの構築およびHIV-1を標的とするRNA干渉法への応用
1. 結果と考察
1-1. HIV-1を標的とするsiRNA設計法の検討
HIV-1やHCVなど多くのRNAウイルスでは,著しく高いゲノム多様性のためにsiRNAの設計が難しいばかりでなく,1種類のsiRNAを単独で用いた場合,そのsiRNAに耐性をもつウイルスが容易に出現しうることが報告されている(Boden et al., 2003a; Das et al., 2004).これらの課題を克服するひとつの方法は,ウイルスの配列のなかで高度に保存された領域を標的とすることである.そこでLos Alamos HIV Sequence Database(http://www.hiv.lanl.gov/)より入手可能なHIV-1グループMのゲノム配列495種(Table S2, 巻末)をもとに,siRNAの標的として有力な候補となりうる高度保存領域を同定した.まず,495種のHIV-1配列を構成する21塩基の部分配列(以下,単に21-merと呼ぶ)をすべて生成し,そのひとつひとつの21-merが495種のうち何種に共通であるかを調べた.たとえばAATAGCAACAGACATACAAACという21-merが495種のうち300種に共通であった場合,その21-merの 保存度(conservation)を300/495×100=60.6 (%)と計算した.ただし,本解析に用いたHIV-1配列の多くは5′ UTR部分の配列を欠いているため,5′ UTR領域の保存度を計算する際に分母を495とすると,保存度が過小に評価されるという問題があった.そこで,5′ UTR領域のうちLTR部分については,各々の配列の3′ LTRの配列を用いて補完した.さらに,保存度の計算では分母を一律に495とするのではなく,保存度を求めようとしている21-merに対応する領域を欠いていない配列の個数をその都度求め,それを分母とした.以上の操作により,5′ UTR領域の21-merについても正確に保存度を評価できた.
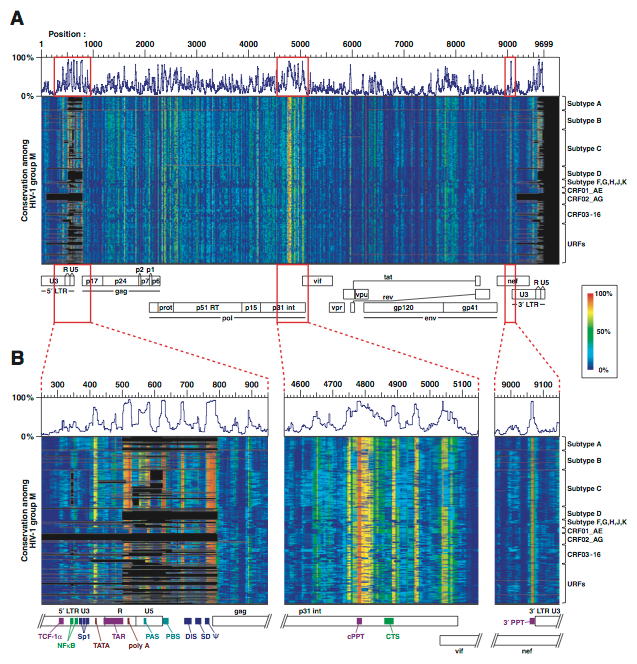
495種のHIV-1配列を構成する21-merは合計で4,417,157種であり,配列の重複を除くと1,208,150種となった.495種のHIV-1配列から生成された全21-merが,重複を除いた際に4分の1程度にしかならない事実は,HIV-1ゲノムの多様性が著しく高いことを示唆した.ついで各21-merの保存度をsiVirusプログラム(Naito et al., 2006)を用いて計算し,青色(保存度0%)から赤色(保存度100%)の点で示したところ(Fig. 2-1A),それらのほとんどが青色に近く,HIV-1ゲノムの大部分の領域は21-merの単位では保存されていないことが明らかになった.具体的には,保存度が70%以上の21-merは全体のわずか1.6%であり,保存度が50%以上の21-merでも全体の5.2%に過ぎなかった(Fig. 2-2).また,すべてのHIV-1配列に共通な,保存度が100%であるような21-merは存在しなかった.これらの結果は,HIV-1を標的とするsiRNAを無作為に設計しても,高い多様性をもつHIV-1を効率よく標的とすることが難しいことを示唆した.なお,HIV-1を標的とするRNAi法の試みは既に複数の研究グループより報告されているが,それらの研究で使用されているsiRNAの多くは保存度が低かった(Table S3, 巻末).
Figure 2-1. HIV-1ゲノムを構成する21-merの保存度.(A) 495種のHIV-1配列から生成した4,417,157種の21-merについて保存度を求め,青色(保存度0%)から赤色(保存度100%)の点で示した.折れ線グラフは各位置における保存度の最大値(最頻値と一致)を示す.(B) 5′ UTR,インテグラーゼ遺伝子内のcPPT/CTS領域,および3′ PPT領域の拡大図.
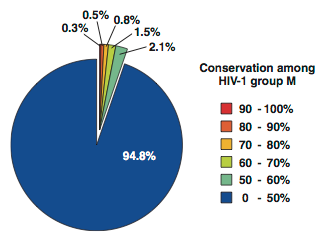
Figure 2-2. 495種のHIV-1配列から生成した全21-merの保存度の分布.
しかしながら,21-merのうち特定の領域に位置するものは高度に保存されていることが判った(Fig. 2-1B).これらの領域の多くはウイルスの遺伝子発現や増殖に必須な配列を含んでいた.すなわち,ウイルスの遺伝子発現に必要なTATA配列やポリAシグナル(AAUAAA),逆転写に必要なprimer activation signal(PAS)およびprimer binding site(PBS),ウイルスのパッケージングに必要なpackaging signal(Ψ),ゲノムの+鎖DNA合成に必要なcentral polypurine tract(cPPT),central termination sequence(CTS)ならびに3′ polypurine tract(3′ PPT)などが高度に保存されていた.これらはすべて,アミノ酸配列ではなくRNAの配列自体もしくはRNAの2次構造を通して機能を発揮するような領域であることは興味ある知見であった.実際,アミノ酸配列のレベルで保存されているような領域の多くは,コドンの3番目の塩基の変異を許容するため,塩基配列レベルでは必ずしも保存されていない例が多かった.
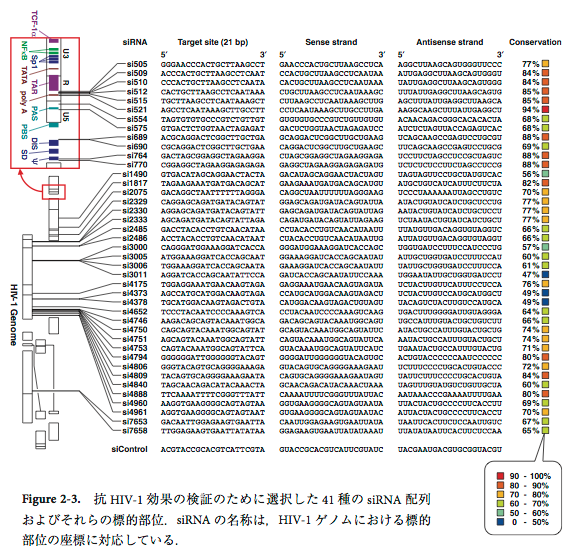
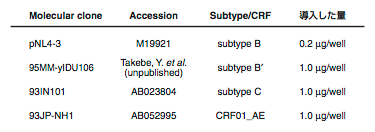
以上の解析により同定した,保存度が70%以上であるような21-merをTable S4(巻末)に示した.この216種のsiRNA配列のうち36種がUi-TeiらのsiRNA設計ガイドラインを満たし,哺乳類細胞で有効と予測された.また,ReynoldsらのsiRNA設計法では30種,Amarzguiouiらの設計法では44種がそれぞれ有効と予測された(Table S4, 巻末).siRNAの抗HIV-1効果を検証するため,Table S4に記載されたUi-Teiらのガイドラインを満たす36種のsiRNAから,感染性分子クローンpNL4-3(GenBank: M19921)と配列が完全一致するものを23種選択し,また,保存度が中程度でUi-Teiらのガイドラインを満たし,かつpNL4-3と完全一致するものを18種選択した.選択した合計41種のsiRNAの配列をFigure 2-3に示した.
Figure 2-3. 抗HIV-1効果の検証のために選択した41種のsiRNA配列およびそれらの標的部位.siRNAの名称は,HIV-1ゲノムにおける標的部位の座標に対応している.
1-2. siRNAの抗HIV-1効果の検証
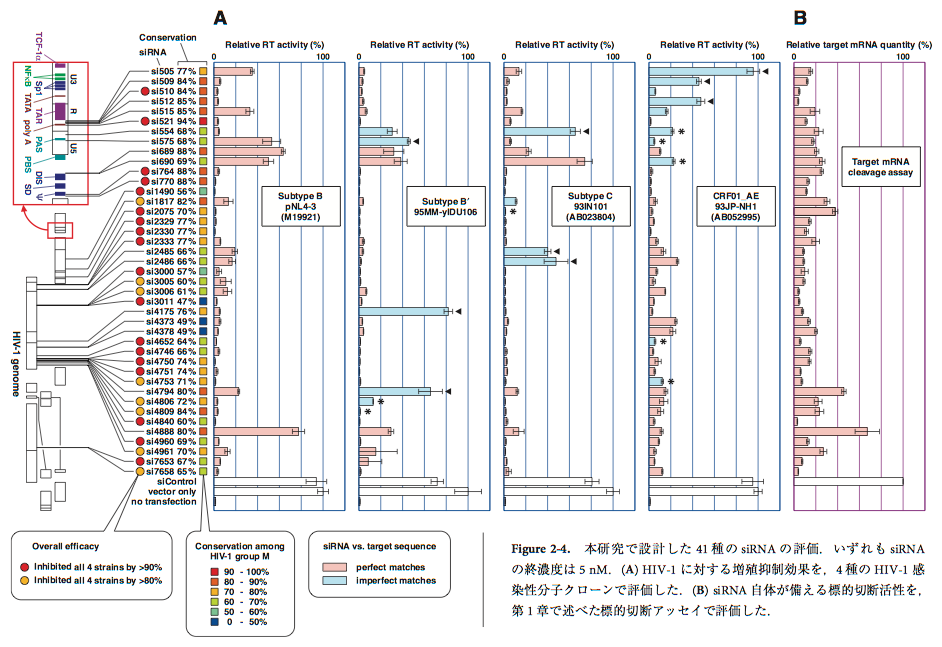
まず,siRNA自体が備える標的切断活性を,第1章で述べたsiRNAの標的切断活性を評価するアッセイ系(以下,標的切断アッセイ)で評価した.すなわち,各siRNAの標的配列をmRNAの一部として発現するコンストラクトを構築し,siRNAとともにHeLa細胞に導入した.ついで,そのコンストラクトから発現させた標的mRNAの分解を,リアルタイムRT-PCR法により定量した.その結果,各siRNAを5 nMで用いた場合,41種のsiRNAのうち39種が標的mRNAを40%以下に抑制した(Fig. 2-4B).si4794とsi4888は有効と予測されたにもかかわらず,5 nMの濃度において十分な抑制を示さなかった.この理由は明らかでないが,si4794は配列中に連続するグアニン残基(GGGGGG)および連続するシトシン残基(CCCCCC)を含むためRNAオリゴの化学合成が難しく,またsi4888は配列中にAAAAUUUUという回文配列が存在し2次構造を取りやすいため,いずれも正しく塩基対を形成した2本鎖siRNAの実効濃度が低下していた可能性がある(Ui-Tei et al., 2004; Reynolds et al., 2004).
Figure 2-4. 本研究で設計した41種のsiRNAの評価.いずれもsiRNAの終濃度は5 nM.(A) HIV-1に対する増殖抑制効果を,4種のHIV-1感染性分子クローンで評価した.(B) siRNA自体が備える標的切断活性を,第1章で述べた標的切断アッセイで評価した.
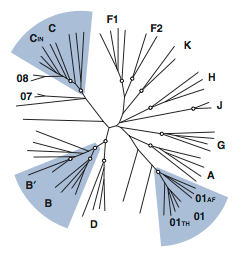
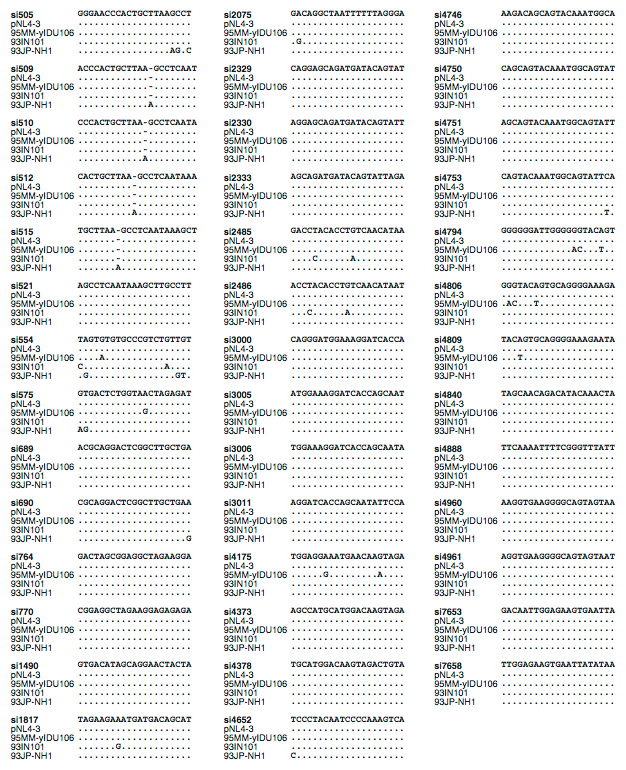
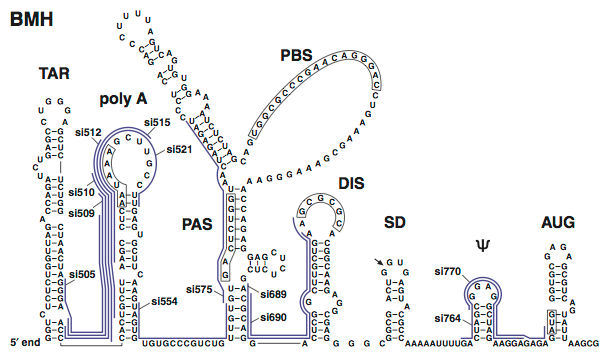
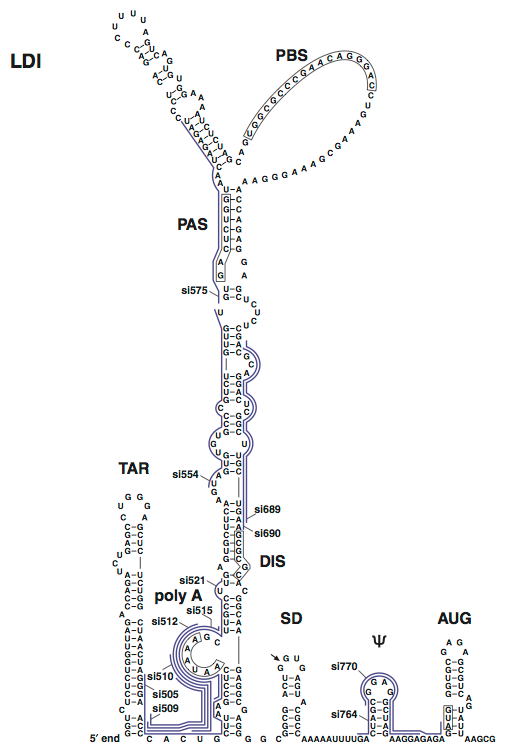
ついで,各siRNAのHIV-1に対する増殖抑制効果を,サブタイプB, C, CRF01_AE属する4種のHIV-1感染性分子クローン(Table 2-2)で評価した.これら3種のサブタイプはアジア地域におけるHIV-1流行の主要な原因となっており,また塩基配列のうえでは相互に隔たりのあるクラスタを形成している(Fig. 2-5, Takebe et al., 2003).本アッセイでは,それぞれの感染性分子クローンをsiRNAとともにHeLa細胞に導入し,48時間後に培地上清を回収してHIV-1由来の逆転写酵素活性を測定することにより,ウイルスの増殖を評価した.その結果,各siRNAを5 nMで導入した場合,41種のsiRNAのうち26種がすべてのHIV-1株の増殖を20%以下に抑制した(Fig. 2-4A, 赤およびオレンジの丸印で表示).残りの15種のsiRNAのうちsi4794/4888を除く13種は,標的切断アッセイにおいて活性が認められ(Fig. 2-4B),またsi690/4794/4888以外の12種は少なくとも1種のHIV-1株の増殖を20%以下に抑制していることから,それらのsiRNA自体は有効であると考えられた.しかしFig. 2-4Aにおいて矢印(◀)で表示したHIV-1株では,siRNA配列と標的配列との間に1塩基〜数塩基のミスマッチが存在するため(Fig. 2-6),それらのHIV-1株の増殖を抑制できなかった.一方,塩基の置換がsiRNAの末端に近い場合にはsiRNAの効果が保持される場合があった(Fig. 2-4A, *で表示).このように末端に近い位置においてミスマッチの影響が少ないという結果は,第1章で述べたオフターゲット効果の解析結果(Fig. 1-10, 1-11, 1-12)とおおむね一致した.またsi689/690については,siRNA配列と標的配列とが完全に一致しているにもかかわらずHIV-1の増殖を抑制できなかった(各感染性分子クローンの標的領域をシークエンスして配列が一致することを確認済み).この領域は,HIV-1の5′ UTRがとりうる2種類の2次構造(Huthoff et al., 2001),すなわちBMH(branched multiple hairpin; Fig. 2-7)とLDI(long distance interaction; Fig. 2-8)の両方において固く閉じており,siRNAが標的配列に接近することを妨げている可能性が示唆された.さらに,si575の効果がpNL4-3と93IN101とで異なる点が注目された.この理由は明らかでないが,ひとつの可能性として,各感染性分子クローンで標的部分の2次構造に差異がありsiRNAの接近のしやすさが異なるのかも知れない.このような2次構造の影響を検討するひとつの方法として,5′ UTR配列の全体をpTRECに挿入したコンストラクトを構築し,標的切断アッセイをおこなう方法が有効と考えられる.
Figure 2-5. HIV-1グループMの分子系統樹(Takebe et al., 2003より作成).
Figure 2-6. 本研究で評価した41種のsiRNA配列と,各感染性分子クローンの標的配列とのアライメント.siRNA配列はアンチセンス鎖の21塩基に対応するセンス側の配列を示した.いずれもRNA分子であるが,塩基配列はGenBankに収録された各データにあわせA, T, G, Cの4文字を用いて記載した.
Figure 2-7. BMI(branched multiple hairpin)型の2次構造.本研究で評価したsiRNAの標的部位を示した.
Figure 2-8. LDI(long distance interaction)型の2次構造.本研究で評価したsiRNAの標的部位を示した.
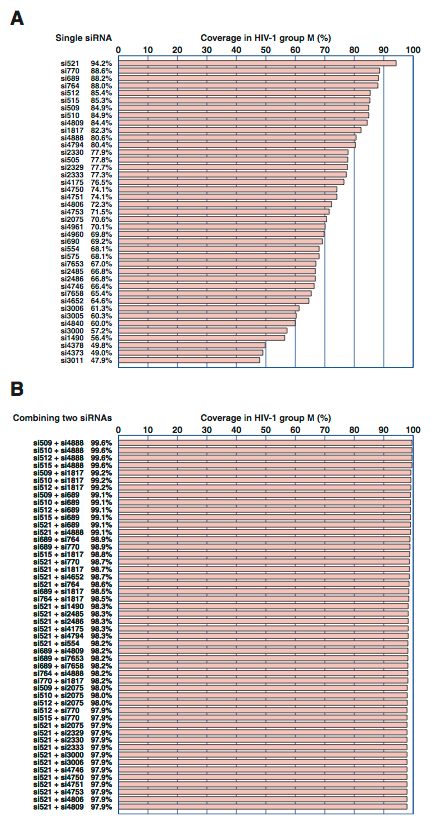
本研究で構築した抗ウイルスsiRNA設計法により,HIV-1の高度な多様性に対応しうるsiRNAを高い確率で設計できた.本研究で実際に評価をおこなったsiRNAのうち,保存度が70%以上であり,かつウイルス増殖を10%以下に抑制できたsiRNAは,
- si521(ポリAシグナルを標的とする; 保存度94%)
- si764/770(Ψ; 88%)
- si510(TAR-ポリAシグナル; 84%)
- si2075(ribosomal slip site; 70%)
- si2329/2330/2333(プロテアーゼ; 77%)
- si4750/4751/4753(インテグラーゼ; 71-74%)
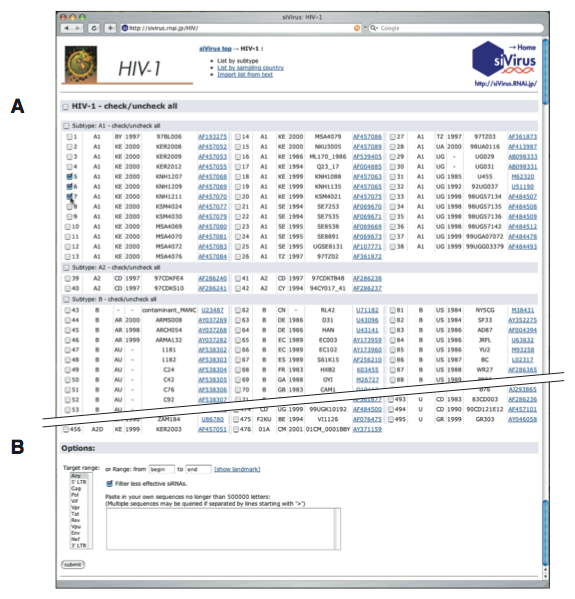
であった.しかしながら,1種のsiRNA単独でデータベースに登録された全HIV-1を標的とすることは不可能であった(Fig. 2-9A).これを克服するひとつの方法は,標的が異なる複数のsiRNAを同時に用いることであろう.本研究で設計し検証をおこなったsiRNAは,2種を組み合わせて同時に用いることにより99%以上のHIV-1に対応可能である(Fig. 2-9B).また本研究で構築した抗ウイルスsiRNA設計法は,HIV-1のみならず,高い多様性をもつHCV,インフルエンザウイルス,SARSコロナウイルス等にも応用できると考えられる.
Figure 2-9. (A) 本研究で評価した41種のsiRNAを単独で使用した場合に標的とすることが可能なHIV-1の割合.各siRNA配列の保存度に等しい.(B) 本研究で評価した41種のsiRNAから2つを選択して同時に使用した場合に標的とすることが可能なHIV-1の割合.
1-3. 抗ウイルスsiRNA設計システムの構築
以上に述べた結果に基づき,(1)ウイルス配列の中で保存度が高く,(2)哺乳類細胞に有効で,(3)オフターゲット効果の少ないsiRNAを設計するシステムを構築した.特に,HIV-1,HCV,A型インフルエンザウイルス,SARSコロナウイルスを標的とする抗ウイルスsiRNA設計システムを,siVirusウェブサーバ(http://siVirus.RNAi.jp/)として公開した(Naito et al., 2006).siVirusウェブサーバの概略は下記のとおりである.
► トップページ(Fig. 2-10)
HIV-1,HCV,A型インフルエンザウイルス,SARSコロナウイルスから,siRNAを設計するウイルスを選択する.各ウイルスの配列はそれぞれ下記のデータベースより入手した.
- Los Alamos HIV Sequence Database(http://www.hiv.lanl.gov/)
- Los Alamos HCV Sequence Database(Kuiken et al., 2005; http://hcv.lanl.gov/)
- NCBI Influenza Virus Sequence Database(http://www.ncbi.nlm.nih.gov/genomes/FLU/FLU.html)
- GenBank(Benson et al., 2007)よりSARSコロナウイルスのゲノム126種を入手.
Figure 2-10. siVirusウェブサーバ(http://siVirus.RNAi.jp/)のトップページ.
► ウイルス配列の選択画面(Fig. 2-11)
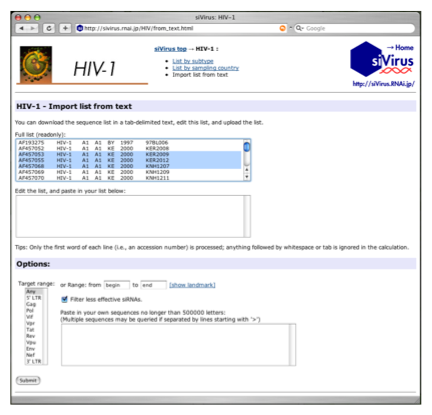
保存度の計算に用いるウイルス配列の母集団を選択する.特定のサブタイプやサンプリング国に絞って保存度を求めることにより,そのサブタイプや地域でもっとも保存度の高いsiRNAを設計できる(Fig. 2-11A).サブタイプや地域を限定する必要のないときは,単にすべての配列を選択してsiRNA設計をおこなってもよい.ウイルス配列の選択は,チェックボックスによるほか,アクセッション番号のリストをテキスト形式で与えることもできる(Fig. 2-12).最下部のオプションでは,siRNAの標的遺伝子または標的とする領域の範囲を指定できるほか,ユーザ独自のウイルス配列をFASTAフォーマットで入力できる(Fig. 2-11B).
Figure 2-11. siVirusによる,HIV-1を標的とするsiRNAの設計画面の例.(A) ウイルス配列の選択画面および (B) オプション指定画面.
Figure 2-12. siVirusによる,HIV-1を標的とするsiRNAの設計画面の例.ウイルス配列の選択は,チェックボックスによるほか,アクセッション番号のリストをテキスト形式で与えることもできる.
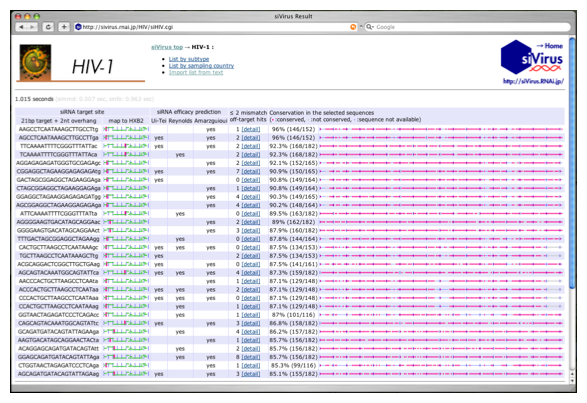
► siRNA設計結果の表示(Fig. 2-13)
ウイルス配列を選択したのちsubmitボタンを押下すれば,直ちに結果が表示される(Fig. 2-13).通常,Ui-Tei,Reynolds,またはAmarzguiouiのsiRNA設計法にしたがう,哺乳類細胞で有効と予測されたsiRNAのみが保存度の高い順番に表示される.結果の表示画面には下記の項目が表示される.
- siRNA target site(siRNAの標的配列およびウイルスゲノムにおける位置)
- siRNA efficacy prediction(哺乳類細胞における有効性の予測)
– Ui-Tei(Ui-Tei et al., 2004のsiRNA設計法を満たす場合にyesと表示)
– Reynolds(Reynolds et al., 2004のsiRNA設計法を満たす場合にyesと表示)
– Amarzguioui(Amarzguioui et al., 2004のsiRNA設計法を満たす場合にyesと表示) - ≤ 2 mismatch off-target hits(2ミスマッチ以内で相同なヒト遺伝子の数)
- Conservation in the selected sequences(選択した配列における保存度)
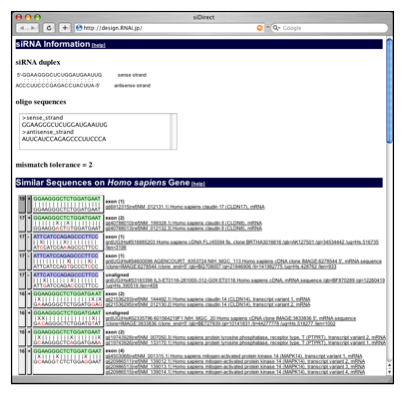
なお,≤ 2 mismatch off-target hitsの[detail]を押下すると,各siRNA配列と3ミスマッチ以内で相同なヒト遺伝子の一覧が表示される(Fig. 2-14).この機能は第1章で述べたsiDirectウェブサーバにより実現している.
Figure 2-13. siVirusによるsiRNAの設計結果.HIV-1のサブタイプB, C, CRF01_AEの合計182種を選択してsiRNA設計をおこなった例.
Figure 2-14. 各siRNA配列と3ミスマッチ以内で相同なヒト遺伝子の一覧.
2. 方法
2-1. siRNA
化学合成のsiRNAは,2本鎖にアニーリングされたものをRNAi社(東京)より購入した.siRNAの配列はFigure 2-3に示した.各siRNAの名称は,HIV-1ゲノム(HXB2; GenBank: K03455)における標的部位の座標に対応している.siControlは,Ui-Teiらのガイドライン(Ui-Tei et al., 2004)を満たし,かつ,ヒトの全転写産物に3ミスマッチ以内で相同な配列が存在しないネガティブコントロールである.
2-2. 細胞の培養およびトランスフェクション
細胞の培養およびトランスフェクションは,第1章の方法にしたがった.
2-3. 標的切断アッセイ
第1章で構築したpTRECベクターのEcoRI部位とXhoI部位との間に,各siRNAの標的配列を挿入した.ベクターに挿入した標的配列部分はすべてシークエンスの確認をおこなった.各コンストラクトの構築に使用したDNAオリゴヌクレオチドの配列をTable 2-1に示した.各コンストラクト(0.5 g/well)をsiRNA(終濃度5 nM)とともにHeLa細胞に導入し,24時間後にRNeasy 96(Qiagen社)で細胞を回収した.その他は第1章の方法にしたがった.
Table 2-1. 抗HIV-1 siRNAの標的切断アッセイのために構築したpTRECコンストラクトの一覧.挿入したDNAオリゴヌクレオチドの配列中,siRNAの標的部分を影付で示した.
2-4. HIV-1逆転写酵素活性の測定
Table 2-2に示した各HIV-1感染性分子クローンをsiRNA(終濃度5 nM)とともにHeLa細胞に導入し,48時間後に培地上清を回収した.ついで,HIV-1に由来する逆転写酵素活性をWilleyらの方法(Willey et al., 1988)にしたがって測定した.
Table 2-2. siRNAの評価に使用したHIV-1感染性分子クローンの概要.
2-5. siVirusウェブサーバの構築
siVirusウェブサーバは,Mac OS Xが稼働するPowerMac G5 Dual 2GHz主記憶6GB(Apple社)1台の上に構築した.ユーザの要求に対して高速に応答できるよう,あらかじめ全ウイルス配列を構成する21-merをすべて生成し,そのひとつひとつの21-merがどのウイルス配列と完全一致するかを求め,データベース化した.このデータベースを参照し,ユーザが選択したウイルス配列の組み合わせにおいて保存度が高い21-merを短時間で求めるプログラム(siVirus core)を作成した.また,2ミスマッチ以内で相同なヒト遺伝子の数を,あらかじめsiDirect(Naito, Yamada et al., 2004)で算出してデータベース化した.
2-6. 共同研究に関する事項
Figure 2-4Bの初期のデータについて,HeLa細胞へのトランスフェクションは東京大学の程 久美子博士がおこなった.
HIV-1の増殖実験および逆転写酵素活性の測定(Fig. 2-4A)は,国立感染症研究所の武部 豊博士,納冨 香子氏,小野木 利成氏がおこない,著者が主体となって分析および検証をおこなった.
siVirus coreの仕様は著者が決定し,実装は東京大学の西川 徹氏がおこなった.なお,ウェブサーバ全体の実装は著者がおこなった.
文献
Trackbacks:0
- Trackback URL for this entry
- /phd/p3/trackback
- Listed below are links to weblogs that reference
- 第二章 from mesoの実験ノート