Home > Archives > 2015-12
2015-12
GGGenomeで塩基配列のモチーフを検索してゲノムブラウザに表示する
- 2015-12-18 (金)
- DBCLS
統合TVの「GGGenome 《ゲゲゲノム》 で転写因子結合サイトを検索してゲノムブラウザに表示する」では、検索結果をBED形式で取得してUCSCゲノムブラウザのcustom trackに登録することにより、GGGenomeのヒットをゲノムブラウザ上に可視化する方法を解説しています。
この記事では、同様のことをもっと簡単にできる方法を紹介します。
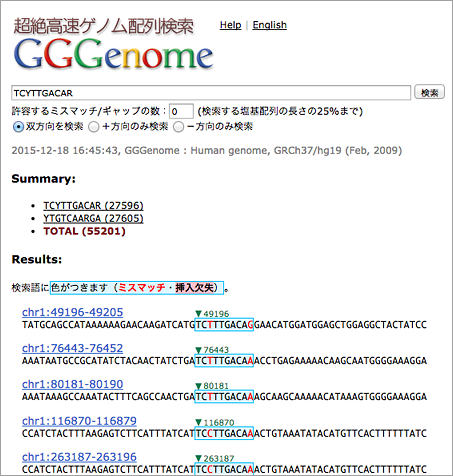
- GGGenomeでヒトゲノムhg19を対象に TCYTTGACAR を検索。55201件のヒットが表示されます。
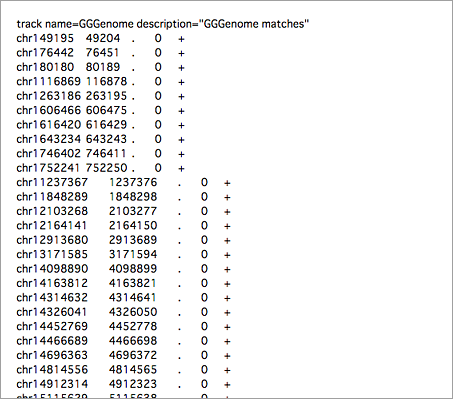
- URLの最後に「.bed」をつけ、BED形式にする。
- このURLをもとに、UCSCゲノムブラウザのURLを作成。
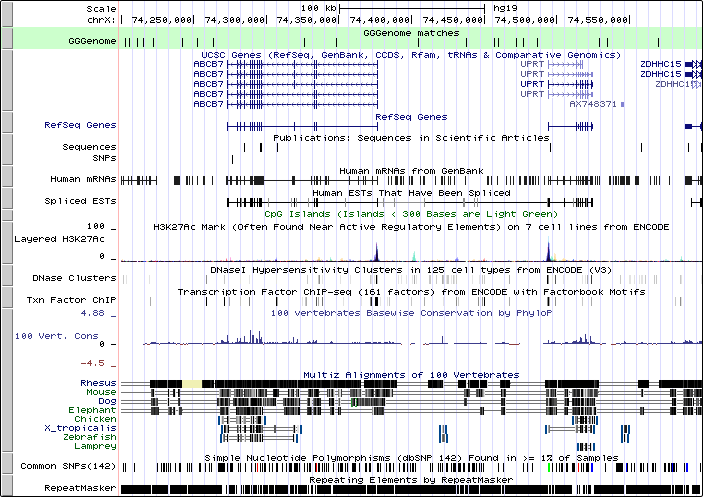
緑色にハイライトされている部分がGGGenomeのヒットです。
このように、URLのなかにBEDデータの場所(http://GGGenome.dbcls.jp/hg19/TCYTTGACAR.bed)を指定するだけで、GGGenomeのヒットを簡単にゲノムブラウザ上に表示することができます。上の例では、表示する領域を position=chrX:74200000-74600000 のように指定していますが、空欄でも問題ありません。
- Comments (Close): 0
- Trackbacks: 0
Home > Archives > 2015-12
- Search
- Feeds
- Meta