- 2014-08-01 (金) 15:46
哺乳類細胞で有効かつ標的遺伝子に特異的なsiRNA設計法の確立とその抗ウイルスRNA干渉法への応用
Antiviral RNA interference based on the development of the highly effective, target-specific siRNA design in mammalian cells
平成18年12月 博士(理学)申請
東京大学大学院理学系研究科 生物化学専攻 内藤 雄樹
・学位論文 (1) – 要旨・緒論(本記事)
・学位論文 (2) – 第一章
・学位論文 (3) – 第二章
・学位論文 (4) – 文献・巻末表・謝辞
Abstract(和文)
RNA干渉(RNA interference; RNAi)とは,細胞に導入した2本鎖RNA(double-stranded RNA; dsRNA)が,相同な塩基配列をもつメッセンジャーRNA(mRNA)を特異的に切断することによって遺伝子発現を抑制する現象である.RNAiの分子機構は,生体内においてはウイルスやトランスポゾンに対する防御機構の一部と考えられているほか,生体内で多数発現しているマイクロRNA(microRNA; miRNA)による遺伝子発現の制御において重要な役割を担っている.また人為的にdsRNAを細胞内に導入することによって,任意の遺伝子を簡便にノックダウンできることから,RNAiは遺伝子の機能解析に有効な手法として注目されている.
哺乳類細胞でRNAi実験をおこなうには,標的遺伝子と相同な,化学合成した約21塩基対の短い2本鎖RNA(short-interfering RNA; siRNA)またはsiRNAを発現するベクターを細胞に導入する方法が一般的に用いられる.しかしsiRNAの配列はどのようなものでも細胞内で機能するわけではなく,無作為に設計したsiRNAの7〜8割はRNAi活性を示さない.さらに,siRNAの長さは約21塩基対と短いため,標的遺伝子とはまったく無関係な遺伝子にも相同部分が存在する確率が高く,siRNAがそのような遺伝子を抑制してしまうことがある(オフターゲット効果).本研究ではまず,特にこれら2つの問題点を考慮した,哺乳類細胞で有効かつオフターゲット効果の少ないsiRNAの設計法を構築した.哺乳類細胞で有効なsiRNAの設計法は,著者らを含む3つの研究グループから提唱されている方法の比較検討をおこない,もっとも良い著者らの方法(Ui-Tei et al. 2004, Nucleic Acids Res. 32, 936-948)を用いた.またオフターゲット効果が少ないsiRNAを設計するために,標的以外のすべての遺伝子に対して相同性が最小となるsiRNA配列を選択するという情報科学的手法を用いた.21塩基の相同性検索は,Yamadaらのアルゴリズム(Yamada et al. 2005, Bioinformatics 21, 1316-1324)を用いた.本研究で構築したsiRNA設計システムによって,ヒトの全遺伝子をほぼ網羅できるゲノムワイドなsiRNA設計をおこなった.
一方,siRNAの標的は内在性遺伝子に限定されず,細胞に感染したウイルス由来のRNAを標的とすることによってウイルスの増殖を抑制できることが示されている.本研究で構築したsiRNA設計法は,哺乳類細胞に感染するウイルスを標的とする場合にもそのまま適用できると考えられる.しかしHIV-1やHCVなど多くのRNAウイルスは,著しくゲノム多様性が高く,1種類のsiRNAを単独で用いた場合,そのsiRNAに耐性をもつ変異ウイルスが容易に出現しうることが報告されている.そこで本研究では,多様性の高いウイルスを対象として,その高度保存領域を同定し,それらの領域を標的とするsiRNAの設計をおこない有効性を評価した.
Abstract(英文)
RNA interference (RNAi) is the process of double-stranded RNA (dsRNA) mediated, post transcriptional gene silencing. It is now widely used to knockdown gene expression in a sequence-specific manner, making it a powerful tool not only for studying gene function, but also for therapeutic applications including antiviral treatments.
In mammalian cells, RNAi can be induced using 21 nucleotide-length short-interfering RNA (siRNA) or siRNA expression vectors. However, the efficacy of each siRNA varies widely depending on its sequence; only a limited fraction of randomly designed siRNAs is functional. Moreover, off-target silencing effects arise when the introduced siRNA has sequence similarities with unrelated genes. In the present study, rational designing of highly functional, off-target minimized siRNAs is proposed. Highly functional siRNAs are selected using novel guidelines that were established through an extensive study of the relationship between siRNA sequences and their RNAi efficacy (Ui-Tei et al. 2004, Nucleic Acids Res. 32, 936-948). Off-target effects are minimized by an exhaustive scan of the non-redundant transcriptome database using accelerated off-target search algorithm developed by Yamada et al. (Yamada et al. 2005, Bioinformatics 21, 1316-1324). The present study also publicizes the entire list of highly effective, off-target minimized siRNA sequences for human and mouse genes listed in the RefSeq database.
siRNA mediated RNAi is also applicable for antiviral therapy by targeting viral sequences. However, in case of RNA viruses, designing functional siRNAs that target viral sequences is problematic because of their extraordinarily high genetic diversity. It is also known that siRNA-resistant viral mutants emerge rapidly due to their high mutation rate. In this study, I attempted to design the effective antiviral siRNAs that target HIV-1. Highly conserved regions within HIV-1 genomes were identified through an exhaustive computational analysis, and the functionality of siRNAs targeting the highest possible conserved regions was validated. Several promising candidates of antiviral siRNAs are presented, which effectively inhibited multiple subtypes of HIV-1 by targeting 5′ UTR, protease, and integrase regions. The present study also publicizes the web-based online software system named siVirus (http://siVirus.RNAi.jp/), which provides functional, off-target minimized siRNAs that target highly conserved regions of divergent viral sequences including HIV, HCV, influenza virus, and SARS coronavirus, all of which pose enormous threats to global human health.
緒論
RNA干渉(RNA interference; RNAi)とは,細胞に導入された2本鎖RNA(double-stranded RNA; dsRNA)が,相同な塩基配列をもつメッセンジャーRNA(mRNA)を特異的に切断することによって遺伝子発現を抑制する現象である.RNAiの分子機構は,生体内においてはウイルスやトランスポゾンに対する防御機構の一部と考えられているほか,生体内で多数発現しているマイクロRNA(microRNA; miRNA)による遺伝子発現の制御において重要な役割を担っている.また人為的にdsRNAを細胞内に導入または発現させることによって,ヒト,マウス,ショウジョウバエ,線虫などを含む多くの真核生物において簡便に任意の遺伝子をノックダウンできることから,RNAiは遺伝子の機能解析に有効な手法として大きな注目を集めている.
RNAiは,1998年にFireらによる線虫を用いた実験ではじめて報告された(Fire et al., 1998).Fireらは,dsRNAを線虫にインジェクションにより導入することで,dsRNAと相同な配列をもつ遺伝子が効率よく抑制されることを示した.一方,そのdsRNAを構成するセンス1本鎖RNAまたはアンチセンス1本鎖RNAを単独で線虫にインジェクションしても,標的遺伝子を抑制する効果は見られなかった.同様の現象は線虫のみならず,ショウジョウバエ(Kennerdell et al., 1998),ゼブラフィッシュ(Wargelius et al., 1999),哺乳類細胞(Ui-Tei et al., 2000)などにも見いだされ,RNAiの基本的な機構が多くの生物種に保存されていることが明らかになった.
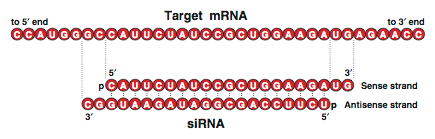
細胞内に導入されたdsRNAは,Dicerによって21〜23塩基対の短い2本鎖RNA(short-interfering RNA; siRNA)に分解される(Bernstein et al., 2001; Ketting et al., 2001).このときsiRNAの5′末端はリン酸基を有し,3′末端は2塩基の突出末端となっている(Fig. 1).siRNAは数種類のタンパク質と複合体(RNA-induced silencing complex; RISC)を形成し,この複合体が標的mRNAを認識し切断する.RISCを構成する主要なタンパクとして,Argonaute(Hammond et al., 2001; Martinez et al., 2002; Doi et al., 2003),Dicer,TRBP(Chendrimada et al., 2005),PACT(Lee et al., 2006)などが報告されている.
Figure 1. siRNAは21塩基対前後の短い2本鎖RNAである.siRNAは5′末端にリン酸基を有し,3′末端は2塩基の突出末端となっている.
RNAiが発見された当初,哺乳類細胞に30塩基対以上の長いdsRNAを導入すると,遺伝子発現を非特異的に抑制するインターフェロン応答が誘導されるため(Grant et al., 1995; Kaufman et al., 1999),哺乳類細胞でRNAiをおこなうことは難しいと考えられていた.しかし2001年にElbashirらによって,約21塩基対のsiRNAを用いればインターフェロン応答を引き起こすことなくRNAiを誘導できることが示されたため,ヒトやマウスを含む哺乳類細胞に対してもRNAiの利用が可能となった(Elbashir et al., 2001a).
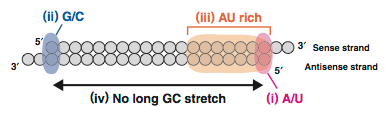
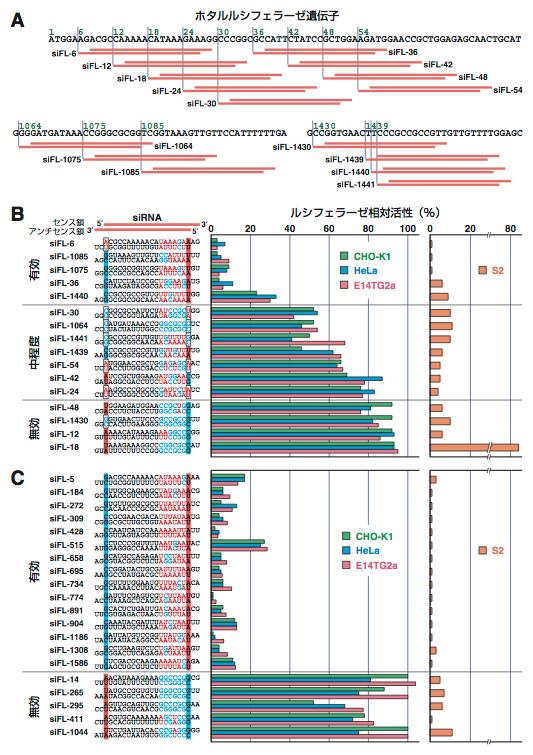
しかしながら,哺乳類細胞に対してsiRNAを用いる場合,siRNAの配列によってRNAi活性に大きな差異が見られることがわかった(Holen et al., 2002).ホタルルシフェラーゼ遺伝子を標的とした実験では,無作為に設計したsiRNAのうちの2〜3割程度しか十分なRNAi活性を示さず,それ以外の多くのsiRNAは機能しなかった(Ui-Tei et al., 2004).そこでUi-Teiおよび著者らは,多数のsiRNAを用いた系統的な解析をおこない,RNAiを効率よく誘導できるsiRNAが示す配列上の規則性を検討した.その結果,(i) siRNAを構成するアンチセンス鎖の5′末端がAまたはUであり,(ii)センス鎖の5′末端がGまたはCであり,(iii)アンチセンス鎖の5′端の7塩基のうち4塩基以上がAまたはUであり,(iv)配列中にG/Cが10塩基以上連続しないようなsiRNA(Fig. 2にi〜ivを図示)は,Figure 3に示すように哺乳類細胞において高い確率で標的遺伝子を抑制できた.一方,(i)〜(iii)の条件をすべて満たさないsiRNAの活性はどれも非常に低かった.この配列規則性は,ヒトおよびマウスの内在性遺伝子を標的とした場合でも,またニワトリ胚における個体レベルの実験でも成り立っていた.さらに,siRNAを細胞に導入する場合だけでなく,DNAベクターからsiRNAを発現させた場合でもこの規則性が同様に成り立っていた.この規則性に基づいてsiRNAを設計することにより,ノックダウンの成功率を飛躍的に高めることが可能となった.
Figure 2. Ui-Teiらによる,哺乳類細胞で高いRNAi活性を示すsiRNA配列の規則性.
Figure 3. ホタルルシフェラーゼ遺伝子に対して設計したsiRNAとそのRNAi効果(Ui-Tei et al. 2004より作成).哺乳類細胞であるチャイニーズハムスターのCHO-K1,ヒトのHeLa,マウスES細胞のE14TG2aにおける結果および,ショウジョウバエのS2細胞における結果を示した.(A) ホタルルシフェラーゼ遺伝子に対して無作為に設計した16種類のsiRNA.(B) 16種類のsiRNAによるRNAi効果を,Dual-luciferase reporter assay system(Promega社)で測定し,RNAi効果の高い順番に並べた.(C) 有効と予測したsiRNA(15種類)と,無効と予測したsiRNA(5種類)のRNAi効果.siRNAの番号は,ホタルルシフェラーゼ遺伝子のコード領域における位置に対応している.
一方,siRNAの長さは約21塩基対と短いため,標的遺伝子とはまったく無関係な遺伝子にも相同部分が存在する確率が高い.そのような場合,siRNAによって標的とは無関係な遺伝子の発現まで抑制される可能性がある.これはsiRNAのオフターゲット効果と呼ばれ,siRNAを用いた遺伝子機能解析や,siRNAを医薬に応用するうえでの深刻な課題のひとつと考えられている(Jackson et al., 2004).オフターゲット効果を回避するためには,標的以外のすべての遺伝子に対して相同性が最小となるsiRNA配列を選択することが望ましい.一般的には,設計したsiRNA配列をBLAST(Altschul et al., 1990)で検索し,無関係な遺伝子がヒットしないものを選択すればよいとされている.しかしBLASTによる検索は,siRNAのような短い配列の検索において見落としが多いという欠点があることが判った.たとえば,Figure 4Aに示すアライメントは19塩基中16塩基が一致しているが,連続した7塩基以上の一致が存在しないためBLASTでは相同性を検出できない.またFigure 4Bのアライメントは19塩基中18塩基が一致しているが,BLASTの標準パラメータは1塩基の一致を+1点,1塩基の不一致を-3点として相同性を評価するため,3′端の3塩基の一致(CTG)を見落とし,15塩基中15塩基の一致という不完全な結果を返す.このようにBLASTは短い配列の相同性検索に不向きであるため,BLASTでsiRNAの特異性を確認する従来の方法ではオフターゲット効果のリスクが過小に評価される(Naito, Yamada et al., 2004).この問題に対しYamadaらは,siRNAのような短い配列の相同性検索を見落としなく高速に実行するアルゴリズムを開発した(Yamada et al., 2005).Yamadaらの方法は,与えられた19塩基の配列に対し,ヒト全遺伝子のなかから3ミスマッチ以内で相同なすべての配列を,数ミリ秒で見落としなく検索可能である.
Figure 4. BLASTは短い配列の相同性を見落とす(Naito, Yamada et al. 2004より作成). (A) 7塩基以上の連続一致が存在しない場合はBLASTで相同性を検出できない.(B) 配列の末端付近にミスマッチがある場合,BLASTは最良のアライメントを見落とす.
本研究では,このようなsiRNA設計において考慮すべき2つの項目,すなわち,(1)哺乳類細胞に対して有効なsiRNAを選択し,かつ,(2)オフターゲット効果が低く特異性の高いsiRNAを選択することが可能なsiRNA設計法の構築をおこなった.第1章では,このsiRNA設計システムの構築について述べ,さらにこのシステムを用いたヒト全遺伝子に対するゲノムワイドなsiRNA設計について論じる.
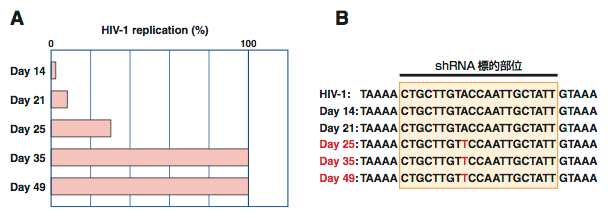
また,siRNAの標的は内在性遺伝子に限定されず,細胞に感染したウイルス由来のRNAを標的とすることによってウイルスの増殖を抑制できることも示されている(Leonard et al., 2006).第1章に述べるsiRNA設計法は,哺乳類細胞に感染するウイルスを標的とする場合にもそのまま適用できると考えられる.しかしながら,HIV-1やHCVなど多くのRNAウイルスでは,著しく高いゲノム多様性のためにsiRNAの設計が難しいばかりでなく,1種類のsiRNAを単独で用いた場合,そのsiRNAに耐性をもつウイルスが容易に出現しうることが報告されている(Boden et al., 2003a; Das et al., 2004).Bodenらの報告によれば,HIV-1のtat遺伝子を標的とするsiRNAと同様の効果をもつヘアピン型RNA(short hairpin RNA; shRNA)を恒常的に発現する細胞にHIV-1を感染させたところ,感染の初期においてはHIV-1の増殖を効率よく抑制できた.しかし感染の25日後には,shRNAの標的部位に1塩基の置換を含むshRNA耐性ウイルスがあらわれた(Fig. 5).このような課題を克服するひとつの方法は,ウイルスの配列のなかで高度に保存された領域を標的とすることであると考えられる.
Figure 5. Boden et al., 2003aより作成.(A) tat遺伝子を標的とするshRNAを恒常的に発現するH9細胞における,HIV-1の増殖抑制効果.HIV-1の増殖はp24抗原をELISAで定量することによって評価した.(B) 培地上清中のHIV-1をシークエンスした結果.25日後よりshRNAの標的部位に1塩基の置換を含む耐性ウイルスがあらわれた.
第2章では,ウイルスの配列のなかで高度に保存された領域を標的に,有効なsiRNAを設計する方法について論じる.本研究ではまず,データベースより入手可能な約500種のHIV-1ゲノム配列を詳細に解析し,siRNAの標的として好適な,21塩基の長さで保存度が高い領域を同定した.ついで,これらの領域を標的とするsiRNAを,第1章で述べるsiRNA設計法に基づき設計した.さらに,設計したsiRNAのHIV-1増殖抑制効果を,複数のサブタイプに属する4種のHIV-1感染性分子クローンで検証した.その結果,多くのsiRNAが4種すべてのHIV-1の増殖を効果的に抑制でき,本研究で構築した抗ウイルスsiRNA設計法の有効性を確認できた.第2章の最後では,本結果に基づく抗ウイルスsiRNA設計システムsiVirusの構築およびウェブサーバの公開についても述べる.
なお,本研究の一部は既に下記に報告した.
-
Ui-Tei, K., Naito, Y. & Saigo, K.
Guidelines for the selection of effective short-interfering RNA sequences for functional genomics.
Methods Mol. Biol. 361, 201-216 (2006). -
Ui-Tei, K., Naito, Y. & Saigo, K.
Essential notes regarding the design of functional siRNAs for efficient mammalian RNAi.
J. Biomed. Biotechnol. 2006, 65052 (2006). -
Naito, Y., Ui-Tei, K., Nishikawa, T., Takebe, Y. & Saigo, K.
siVirus: web-based antiviral siRNA design software for highly divergent viral sequences.
Nucleic Acids Res. 34, W448-W450 (2006). -
Naito, Y., Yamada, T., Matsumiya, T., Ui-Tei, K., Saigo, K. & Morishita, S.
dsCheck: highly sensitive off-target search software for double-stranded RNA-mediated RNA interference.
Nucleic Acids Res. 33, W589-W591 (2005). -
Naito, Y., Yamada, T., Ui-Tei, K., Morishita, S. & Saigo, K.
siDirect: highly effective, target-specific siRNA design software for mammalian RNA interference.
Nucleic Acids Res. 32, W124-W129 (2004). -
Ui-Tei, K., Ueda, R., Zenno, S., Takahashi, F., Doi, N., Naito, Y., Yamamoto, M., Hashimoto, N., Takahashi, K., Hamada, T., Tokunaga, T. & Saigo, K.
RNA interference induced by transient or stable expression of hairpin structures of double-stranded RNA in Drosophila and mammalian cells.
Mol. Biol. 38, 276-287 (2004). -
Ui-Tei, K., Naito, Y., Takahashi, F., Haraguchi, T., Ohki-Hamazaki, H., Juni, A., Ueda, R. & Saigo, K.
Guidelines for the selection of highly effective siRNA sequences for mammalian and chick RNA interference.
Nucleic Acids Res. 32, 936-948 (2004).
第一章 哺乳類細胞で有効かつ標的遺伝子に特異的なsiRNA設計法の構築
Trackbacks:0
- Trackback URL for this entry
- /phd/p1/trackback
- Listed below are links to weblogs that reference
- 要旨・緒論 from mesoの実験ノート