- 2014-08-01 (金) 15:46
(執筆:2005年11月)
本稿は、筆者が東京大学大学院理学系研究科 生物化学専攻 西郷研究室に所属していた頃に執筆した下記の記事をもとにまとめたものです。
- 内藤 雄樹、山田 智之、程 久美子、森下 真一、西郷 薫 (2005)「siRNAの選択法」バイオテクノロジージャーナル 2006年1-2月号、23-28、羊土社
概要
哺乳類細胞でRNAi実験を行うためには、標的遺伝子と相同な約21塩基対の短い二本鎖RNA (siRNA) またはsiRNAを発現するベクターを細胞に導入する方法が一般的に用いられる。しかしsiRNAの配列はどこでもよいというわけではなく、無作為に選択したsiRNAの多くはRNAi効果を示さない。本稿では、哺乳類細胞で十分なRNAi効果が期待でき、標的遺伝子に対する特異性が高いsiRNAの設計法について紹介する。
はじめに
哺乳類細胞ではsiRNAの配列によってRNAi効果が大きく異なるため、適当にsiRNAを設計しても目的の遺伝子を十分に抑制できないことが多い。しかも化学合成のsiRNAは高価なため、数多く注文して効くものだけを採用するような実験計画は立てにくい。そのため、標的遺伝子全体に対する二本鎖RNAをin vitro転写により合成し、二本鎖RNA切断酵素によりランダムに切断した、さまざまな配列のsiRNA混合物を利用すればよいとする方法もある (文献1)。一方で、われわれも含めたいくつかのグループから、実験的な根拠に基づき、有効と考えられるsiRNAの配列を設計するアルゴリズムが提唱されている。本稿ではまず、われわれの方法を中心に、「効くsiRNA」を設計する方法について紹介し、さらに、siRNAが標的とは無関係な遺伝子を意図せず抑制してしまう「オフターゲット効果」を回避するための方法を概説する。最後に、これらの煩雑なsiRNA設計の作業を簡単に行えるウェブサーバを構築しているので紹介したい。
効くsiRNAの設計法
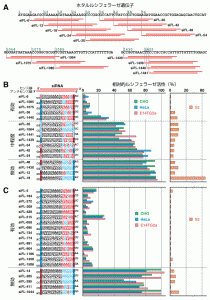
われわれは、ホタルルシフェラーゼ遺伝子に対する16種類のsiRNAを無作為に設計し、そのRNAi効果をホタルルシフェラーゼ活性を指標として種々の培養細胞で測定した (図1A)。ショウジョウバエの培養細胞S2では、設計した16種のsiRNAのうち15種が効果的であった。しかし哺乳類細胞では、チャイニーズハムスターのCHO-K1、ヒトのHeLa細胞、マウスES細胞であるE14TG2a細胞のすべてにおいて、わずか5種のみが有効であり、それ以外の11種では十分な抑制効果が認められなかった (図1B)。このように哺乳類細胞では、標的遺伝子に対して無作為にsiRNAを設計しても3割程度のsiRNAしか効かず、それ以外の多くのsiRNAは標的遺伝子を十分に抑制できないことがわかった。このことは哺乳類細胞を対象とするRNAi解析の大きな障害のひとつと考えられた。
図1:ホタルルシフェラーゼ遺伝子に対して設計したsiRNAとそのRNAi効果。A:ホタルルシフェラーゼ遺伝子に対して無作為に設計した16種類のsiRNA。B:16種類のsiRNAによるRNAi効果を、Dual-Luciferase Reporter Assay System (Promega) で測定し、RNAi効果の高い順番に並べた。C:有効と予想したsiRNA (15種類) と、無効と予想したsiRNA (5種類) のRNAi効果。siRNAの番号は、ホタルルシフェラーゼ遺伝子のコード領域における位置を示している。
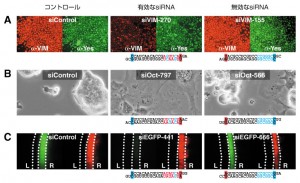
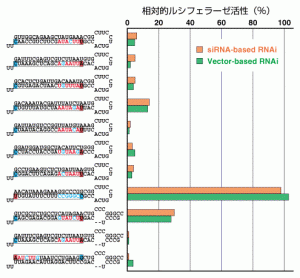
哺乳類細胞においてRNAi効果の高い順番にsiRNAを並べてみると、効くsiRNAと効かないsiRNAの配列には対称的なパターンが存在することがわかった (図1B)。すなわち効くsiRNAは、(1) アンチセンス鎖 (語句説明) の5′末端がAまたはU であり、(2) センス鎖の5′末端がGまたはC であった。また、(3) アンチセンス鎖の5′の7塩基中の4塩基以上はAまたはU である点が共通であった。一方、効かないsiRNAは(1)~(3)の条件をすべて満たさないという全く対称的な特性を示していた。そこで上記の3つの条件を満たすsiRNAが効くかどうかを検証するために、ホタルルシフェラーゼ遺伝子に対して、(1)~(3)をすべて満たす、効くと予想されるsiRNAを無作為に15種類、これらとは対称的な、効かないと予想されるsiRNAを5種類設計し、RNAi効果を調べた (図1C)。効くと予想されたsiRNAはすべて高いRNAi効果を示し、効かないと予想されたsiRNAのRNAi効果は非常に低いことが確認できた。また内在性遺伝子に対しても同様に検討したところ、ヒト中間系フィラメントであるビメンチン (図2A)、マウスES細胞の分化に関わる転写因子のOct-4 (図2B) などのヒト・マウス内在性遺伝子においても、上記3つの条件を同時に満たす、効くと予想されたsiRNAは、非常に効率よくRNAiを誘導することがわかった。また、ニワトリ胚における個体レベルでのRNAi実験においても、上記の3つの条件を満たすsiRNAは有効であった (図2C)。しかし、効かないと予想されたsiRNAは、どの場合もほとんど無効であった。これらの結果より、上記(1)~(3)の3つの条件を満たすsiRNAは、内在性遺伝子に対しても、また個体レベルでも、効率的にRNAiを誘導できることがわかった。さらに、このsiRNA設計の条件はsiRNAを用いてRNAiを行うときだけでなく、DNAベクターによってsiRNAを発現させる際も同様に有効であった (図3)。
| 語句説明:siRNAのアンチセンス鎖 – siRNAを構成する二本鎖のうち標的mRNAと相補的な鎖で、ガイド鎖とも呼ばれる。こちらの鎖が取り込まれたRISCが標的mRNAを認識・切断すると考えられる。 |
図2:有効なsiRNA設計法の内在性遺伝子および個体レベルでの検証。A:ヒトHeLa細胞におけるビメンチン遺伝子に対する検証。ビメンチンsiRNA (50nM) を3日連続トランスフェクションし、最後のトランスフェクションの翌日に、ビメンチン抗体およびコントロールとしてのYes抗体で染色した。siVIM-270は有効と予想したsiRNA。siVIM-155は無効と予想したsiRNA。siVIM-270でビメンチンの発現が抑制されている。B:マウスES細胞におけるOct-4遺伝子に対する検証。ES細胞は、Oct-4の発現量の減少によって栄養外胚葉細胞に分化する。siOct-797は有効と予想したsiRNA。siOct-566は無効と予想したsiRNA。siOct-797で栄養外胚葉細胞に分化している。C:ニワトリ個体における検証。pCAGGS-EGFPとpCAGGS-DsRedを、EGFPに対するsiRNAと同時にエレクトロポレーションによってニワトリ2日目胚の脊髄の右側半分に導入し、4日目胚でRNAi効果を検討した。siEGFP-441は有効と予想したsiRNA。siEGFP-666は無効と予想したsiRNA。siEGFP-441でEGFPの発現が抑制されている。
図3:siRNAによるRNAi効果とDNAベクターを用いたRNAi効果の比較。DNAベクターから転写されると考えられるshRNAの構造を左に示す。siRNAによるRNAi効果は、siRNAを細胞に導入して1日後に測定し、DNAベクターによるRNAi効果は3日後に測定しているが、配列ごとにほぼ一致している。
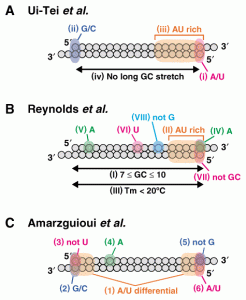
しかし、上記3つの条件を満たしていても、最初に検討した16種のsiRNAのうち、siFL-1439だけはRNAi効果が低かった (図1B)。詳細な解析を行った結果、siRNAの二本鎖領域中にあるG/Cの連続は少ない方がよく、siFL-1440のように9個まではRNAi効果が高いという範疇に入るが、10個以上 (siFL-1439) になると極端にRNAi効果が低下することがわかった。そこで前述の3つの条件に、(4) G/Cの連続が9個以下、という4つ目の条件を加えて、効くsiRNA設計のためのガイドラインとした (図4A; 文献2)。これまでにわれわれは、効くsiRNA設計のための4つのガイドラインを満たす100個以上のsiRNAを設計してそのRNAi効果を検討したが、ほとんどのsiRNAで高いRNAi効果が認められている。しかし一方で、4つの条件を同時に満たしていなくても効くsiRNAがあることから、このガイドラインはすべての効くsiRNAを網羅的に特定するものではないと考えている。
われわれ以外のグループからも、効くsiRNAの設計法について実験結果に基づいた具体的なガイドラインがいくつか提唱されている (図4B,C; 文献3,4)。これらのsiRNA設計法は、いくつかの相違点があるものの、アンチセンス鎖の5′末端はAまたはUであることが好ましく、アンチセンス鎖の5′末端からある程度の長さ (3~7塩基) にAまたはUが多いほうが有効である点で一致している。さらに、センス鎖の5′末端は、GまたはCであるほうが好ましいようである。
図4:効くsiRNAの配列設計法。A:われわれの提唱する効くsiRNA設計のガイドライン。B:Reynoldsらによる、効くsiRNAの設計法。C:Amarzguiouiらによる、効くsiRNAの設計法。
RNAiのプロセスにおいて、siRNAはRISC (語句説明) と呼ばれる複合体に取り込まれるが、活性型のRISCにはsiRNAの二本鎖のうちの1本のみが取り込まれている。Schwarzらは、siRNAの両末端の塩基対形成の強さに注目し、siRNAのセンス鎖とアンチセンス鎖のうち、どちらの鎖が活性型RISCに取り込まれているかを解析した。その結果、siRNAのアンチセンス鎖の5′末端側 (本稿の図でsiRNAの右側) が、センス鎖の5′末端側 (同じく左側) よりも塩基対形成が弱い場合には、siRNAのアンチセンス鎖が主にRISCに取り込まれ、したがって効率よくRNAiが誘導されると報告している (文献5)。またKhvorovaらは、RNAi効果の高いsiRNAは、アンチセンス鎖の5′末端部が熱力学的に安定性が低いことを示しており、このことがsiRNAの二本鎖の巻き戻しの方向性と、活性型RISCにどちらの鎖が取り込まれるかを決定すると報告している (文献6)。これらの報告は、効くsiRNAの設計にはアンチセンス鎖の5′末端および末端から一定の部分にAUが多く、センス鎖の5′末端がGまたはCであるものを選択するのが有効であることとよく一致している。
| 語句説明:RISC – RNA-induced silencing complex。このsiRNA-タンパク質複合体が標的mRNAを認識・切断することによって標的遺伝子の発現が抑制される。 |
siRNAのオフターゲット効果とその対策
siRNAの長さは約21塩基と短いため、標的遺伝子とは全く無関係な遺伝子にも相同部分が存在する確率が高い。そのような場合、siRNAによって標的とは無関係な遺伝子の発現まで抑制される可能性がある。この現象はsiRNAのオフターゲット効果と呼ばれており、siRNAを用いた遺伝子機能解析や、siRNAを医薬に応用するうえで深刻な問題のひとつとなっている (文献7)。
オフターゲット効果を回避するためには、標的以外のすべての遺伝子に対して配列類似性が最小となるsiRNA配列を選択することが望ましい。一般的には、設計したsiRNA配列をBLAST (語句説明) で検索し、無関係な遺伝子がヒットしないものを選択すればよいとされている。しかし、ひとつひとつのsiRNA配列をBLAST検索するのは時間と労力がかかるうえ、siRNAのような短い配列の検索では見落としが多いという重大な欠点がある。このためBLASTでsiRNAの特異性を確認する従来の方法では、オフターゲット効果のリスクを過小に評価してしまうことになる。そこでわれわれは、siRNAのような短い配列の類似性検索を高速かつ確実に実行できるプログラムを開発し、後述するsiRNA設計ウェブサーバ「siDirect」に取り込んでいる (文献8,9)。そのため、設計したsiRNA配列と3ミスマッチ以内で相同な遺伝子を見落としなく検索することが可能となっている。具体的な使用方法については次の節で紹介する。
| 語句説明:BLAST – 代表的な配列類似性検索プログラム。まず7~11塩基程度の短い完全マッチを探し、それを頼りに左右にアライメントを伸長していく。7~11塩基の完全マッチが存在しない場合や、siRNAの末端近くにミスマッチがある場合に見落としが生じる (文献8,9)。 |
このように、標的以外の遺伝子に対するオフターゲット効果の可能性は、配列上のミスマッチから推定することもできるが、実験的にも判断する方法がいくつかある。例えば、同一の標的遺伝子に対して2種のsiRNAを設計し、同じ結果となることを確認する。また、オフターゲット効果の可能性のある遺伝子については、その発現量の変化をRT-PCRなどによって確認する。さらに、標的とする遺伝子の発現コンストラクトが作製可能な場合は、siRNAの標的部分にアミノ酸配列が変わらないような変異を導入し、それを用いてレスキューされることを確認する方法も有効といえる。
siRNA設計ウェブサーバの活用
これまでの節で述べたように、siRNAを設計するためには少なくとも、
- 哺乳類細胞で効く配列
- オフターゲット効果の低い配列
という2つの基準を満たす配列を選択することが求められるが、これを手作業で行うのは困難である。そこでわれわれは、東京大学の森下研究室と共同で、上記の2つの基準を満たすsiRNAを簡単に設計するためのウェブサイト「siDirect」(http://design.RNAi.jp/) を公開している (文献8)。
- リンク – 森下研究室 (東京大学大学院新領域創成科学研究科 情報生命科学専攻)
siDirectのトップページを図5Aに示すが、基本的にはこの画面上に標的遺伝子の塩基配列またはAccession番号を入力するだけで、効くsiRNA設計のガイドラインを満たす配列のリストが得られる (図5B)。さらに、上部のリストおよび青色のバーで表示されたsiRNAは、センス鎖・アンチセンス鎖とも、本来の標的以外のあらゆる配列に対して必ず3塩基以上のミスマッチが存在する配列である。ヒトのRefSeq mRNAデータベースにおいては、このようなsiRNA配列は全体の約10%存在するが、4塩基以上のミスマッチを保証できるものはほとんど存在しない (文献8)。したがって、siRNA設計においては3塩基以上のミスマッチがある配列を選択するのが最善といえる。このようなsiRNAがひとつも設計できない場合は、2番目のリストおよび水色のバーで表示されたsiRNAを選択すればよい。これらのsiRNAは、少なくともsiRNAのアンチセンス鎖については、すべてのオフターゲット配列に対して必ず3塩基以上ミスマッチが存在する。この場合、siRNAのセンス鎖側の特異性は保証できないが、もしセンス鎖と相同なオフターゲット遺伝子が存在しても、センス鎖側は効率良くRISCに取り込まれない可能性が高く、その遺伝子に対するオフターゲット効果はおそらく問題にならないと考えられる。
図5Bの画面に表示されているsiRNA配列をクリックすると、そのsiRNA配列と3ミスマッチ以内で相同な遺伝子がすべて表示され、設計したsiRNAの特異性を確認できる (図5C)。siRNAのアンチセンス鎖 (緑) およびセンス鎖 (青) との相同部分のアライメントが表示される。
より詳しい設定を行いたい場合には、最初の画面でGC含量や独自の配列ルールなど、必要に応じて各種のオプションを指定できる。
図5:siRNA設計ウェブサーバ、siDirect (http://design.RNAi.jp/)
参考文献
- Kittler R et al., Nat. Methods 2, 779-784 (2005). [PubMed]
- Ui-Tei K et al., Nucleic Acids Res. 32, 936-948 (2004). [FullText]
- Reynolds A et al., Nat. Biotechnol. 22, 326-330 (2004). [FullText]
- Amarzguioui M & Prydz H, Biochem. Biophys. Res. Commun. 316, 1050-1058 (2004). [PubMed]
- Schwarz DS et al., Cell 115, 199-208 (2003). [PubMed]
- Khvorova A et al., Cell 115, 209-216 (2003). [PubMed]
- Jackson AL & Linsley PS, Trends Genet. 20, 521-524 (2004). [PubMed]
- Naito Y, Yamada T et al., Nucleic Acids Res. 32, W124-W129 (2004). [FullText]
- Yamada T & Morishita S, Bioinformatics 21, 1316-1324 (2005). [FullText]
Trackbacks:0
- Trackback URL for this entry
- /sirna_design/trackback
- Listed below are links to weblogs that reference
- siRNA設計法 ~RNAi効果が高く標的に特異的な配列~ from mesoの実験ノート