- 2013-06-25 (火) 12:55
- DBCLS
6/24(月)〜28(金)はDBCLSにて国際開発者会議 BioHackathon 2013 が行われています。この期間にmesoがやりたいことをまとめました。
【GGRNA】GGRNA 2.0 リリース&ソース公開
現在の GGRNA は、圧縮接尾辞配列のインデックスをメモリに載せて検索しているため、メモリ容量の制約から13種類のモデル生物に限ってサービスを提供してきました。また、ヒット件数が極端に多い場合は圧縮接尾辞配列の解凍にすごく時間がかかるという弱点がありました。そこで、内部の検索エンジンを最新版のSedueに置き換えて、(非圧縮の)接尾辞配列のインデックスをSSDに載せる方針(以下、SSD方式)に変更することを計画しています。
SSD方式のメリットは、データベースの容量の制約がなくなることです。たとえば約3TBのSSD領域を用意すれば、GenBank/EMBL/DDBJ国際塩基配列データベースに登録されたゲノム以外の全ての配列を全文検索できるようになります(実はもうやっていてテスト段階)。また、ヒット件数が極端に多い場合でも素早く結果を返せるようになります。
逆にデメリットは、現在のGGRNAで提供している機能のうち、ミスマッチを含む塩基配列の検索ができなくなることと、塩基配列やアミノ酸配列の hit position が取得できなくなること。配列検索にこだわっているGGRNAとしては、このダウングレードは痛手なので、なかなかSSD方式への移行を決断できませんでした。
いろいろと悩んだ結果、ミスマッチを含む塩基配列の検索は GGGenome《ゲゲゲノム》という配列検索に特化したサービスに移してしまうことにしました。また、PFI社の大野さんをはじめとするメンバーがSedueのhit position取得プラグインを作成中で、これが完成すればSSD方式に移行できそうです。
7月には統合データベースのユーザ評価が比較的大規模に行われる予定で、その対象サービスのひとつにGGRNAを入れて頂いたので、多くの方のフィードバックをいただけるチャンスです。それまでにSSD方式への移行を済ませて GGRNA 2.0 をリリースしたいと考えています。
さらに、GGRNA 2.0 からはソースを公開する予定です。実はGGRNAの論文を査読してくださったreviewerのひとりが、オープンソースでやったらどうかとコメントしてくれたのですが、ぜひそうしたいと返答してからかなり時間が経ってしまいました(やるやるサギ)。このreviewerはたぶん国内の知っている方で、とても丁寧に査読をしてくれて多くのconstructiveなコメントを頂き感動したのですが、そのときの約束を守れていないので顔向けできない状況なのです(笑
(8/26更新)
7/8に GGRNA ver.2をリリース しました。
【GGRNA/GGGenome】Galaxyと連携
GGRNA / GGGenome の基本的な検索機能はほぼ完成しているので、他のツールとの連携を強化したいと思っていたところ、@32nm さんからこんな連絡をいただきました。
@meso_cacase GGRNA にはGalaxy のGet Data/UCSC Main table browserのように検索結果をSend query to Galaxyする機能を実装する予定はありすか?特に配列検索でData Exportのタブ切り相当で返るように (Twitter: 2013/5/29 – 0:33)
それは良さそうですね、と返信すると参考文献を教えてくださいました。
@meso_cacase どもども。http://database.oxfordjournals.org/content/2011/bar011.full が参考になるはずです〜 (Twitter: 2013/5/29 – 22:42)
【GGGenome】検索結果のうち特定のフィールドだけを返すREST API
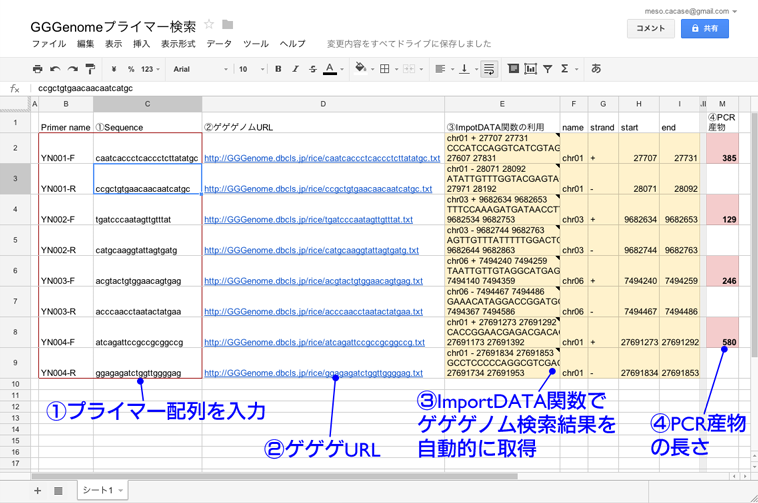
GGGenome は検索クエリがURLと1対1対応しており、しかもURLの末尾に .txt をつけると検索結果をタブ区切りテキストで取得できます(例: http://GGGenome.dbcls.jp/TTCATTGACAACATT.txt)。この機能を利用すると、スプレッドシートにプライマー配列をコピペするだけで、自動的にGGGenome検索をしてPCR産物の長さや増幅する場所を求めることができます(図)。このとき、GGGenomeの検索結果のうち特定のフィールド(例えば start position)だけを取得できる手段があるとさらに便利なので、TogoWS のURL規則を参考に実装したいです。
下の図では、Google Driveの関数 ImportDATA( URL ) を利用してGGGenomeの結果を直接セルにインポートしてみました。「①プライマー配列を入力」のところだけ入力すると、残りの部分は自動的に埋まります。2つのプライマー間で染色体上の座標を引き算すれば、PCR産物の長さも計算できます。
(6/27更新)
上記の Googleスプレッドシート を読み出し専用で公開しました。ImportDATA 関数によるGoogleスプレッドシートとGGGenomeの連携法は、@hkanekane さんから教えていただきました。
【GGGenome】TogoGenomeと連携して遺伝子名表示
GGGenome で塩基配列を検索すると染色体上の座標が返ってきますが、TogoGenome と連携してその位置にある遺伝子名なども表示できれば便利になるはず。
【BioRuby】siRNA設計ライブラリのアップデート
BioRubyには二階堂さん作成のsiRNA設計ライブラリ(sirna.rb)があり、そのなかで Ui-Teiらの設計法とReynoldsらの設計法(リンク先のFig. 1-1を参照)が実装されています。mesoは前者を発表したチームの一人で(Ui-Tei et al., Nucleic Acids Res. 2004)、sirna.rb のコードを読んでみると各ruleの解釈が原著論文の意図と少し違う部分があることに気づいたので、下記のようにアップデートのリクエストをしようと思っています:
ハッカソンではBioRubyのメジャーアップデートが予定されているそうなので、それにあわせて採用してもらえるとありがたいです。
(6/27更新)
ユニットテスト(test_sirna.rb)も修正し、コミットメッセージを詳しく書いてpull requestすればよいとのこと。
(8/26更新)
下記のように、pull requestを取り込んでいただきました。
- Newer: GGRNA ver.2リリース
- Older: difff《デュフフ》リニューアル
Trackbacks:0
- Trackback URL for this entry
- /archives/2417/trackback
- Listed below are links to weblogs that reference
- BioHackathon 2013でやりたいこと from mesoの実験ノート