mesoの実験ノート
統合遺伝子検索GGRNA
- 2011-06-27 (月)
- DBCLS
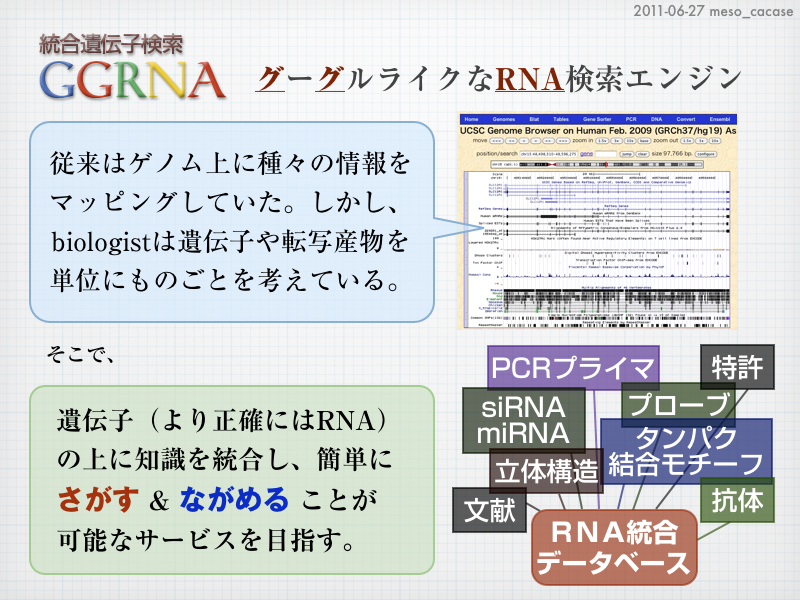
mesoが構築している「遺伝子をGoogleのように(?)検索できるサイト」の名前を「GGRNA」にしてみました。表向きはグーグルライクなRNA検索エンジンでGGRNA。しかし裏では “ググるな” と呼んでいたりします。
これに伴いURLも変更しました。今までのURLも生きていますが、今後はこちらをお使いください。
- 統合遺伝子検索GGRNA —
http://bit.ly/GGRNA→ http://GGRNA.dbcls.jp/ (8/18更新:URLを変更)
下記は定例会議で報告した資料の一部。研究総括からは「Googleに怒られないか?」というご指摘が(爆 もし怒られたら変えます。。。
資料の2枚目は今後の方針について。GGRNAは遺伝子を「さがす」仕組みを実装したもの。それと同じくらい大切なのが、ヒットした遺伝子を「ながめる」仕組み。短い配列や立体構造、文献などのさまざまな知識をRNAのうえに整理して見せたいのですが、その見せ方の部分を工夫したいと思っており、ある程度形になったらここでご紹介します。
- Comments (Close): 0
- Trackbacks: 0
siDirectで大量のsiRNAを設計する
- 2011-06-20 (月)
- DBCLS
mesoが東大生化で開発していたsiRNA設計サーバ・siDirectを使って大量にsiRNAを設計したいというリクエストをいただいた。siDirectは、RNAi活性が高く(つまり良く効く)、標的以外の遺伝子が意図せず抑制される「オフターゲット効果」が少ないsiRNAを効率よく設計できるウェブサーバである。
- siDirect 2.0 — http://siDirect2.RNAi.jp/
- @_junk_0 さん作の siDirect 2.0 解説動画
実はそういう要望は以前からときどき頂いていたので、この機会にsiDirectのウェブをcrawlするスクリプトを書いてみた。
- siDirect2crawl.pl (gist.github)
つかいかた:
% ./siDirect2crawl.pl sequence.txt
sequence.txtは、FASTAファイルもしくは塩基配列だけを格納したテキストファイル。ATGCU以外の文字は無視(削除)される。大文字小文字は区別しないが、結果のtarget sequenceの欄は入力ファイルの大文字小文字が反映される。WWW::Mechanizeというモジュールを使用しているので、あらかじめCPANからインストールする必要がある。
結果の例:
[siDirect v.2.0 | 2011-06-20 18:07:26]
target position target sequence RNA oligo, guide passenger functional siRNA selection seed-duplex stabilty (Tm), guide passenger min. number of mismatches against off-targets, guide passenger
24-46 cagaagaatggtacaaatccaag UGGAUUUGUACCAUUCUUCUG GAAGAAUGGUACAAAUCCAAG U 20.1 12.0 2 2
159-181 cccttaaaggaaccaatgagtcc ACUCAUUGGUUCCUUUAAGGG CUUAAAGGAACCAAUGAGUCC U 18.1 11.0 2 2
261-283 aggatgagattcagaatatgaag UCAUAUUCUGAAUCUCAUCCU GAUGAGAUUCAGAAUAUGAAG U 8.7 20.4 2 3
461-483 ctggttgatacccactcaaaaag UUUUGAGUGGGUAUCAACCAG GGUUGAUACCCACUCAAAAAG U 19.2 16.1 3 2
462-484 tggttgatacccactcaaaaagg UUUUUGAGUGGGUAUCAACCA GUUGAUACCCACUCAAAAAGG U 12.2 16.1 3 2
491-513 ctgattaagacggttgaaactag AGUUUCAACCGUCUUAAUCAG GAUUAAGACGGUUGAAACUAG U 14.9 6.9 3 3
519-541 gacaggttatcaacgaaacttct AAGUUUCGUUGAUAACCUGUC CAGGUUAUCAACGAAACUUCU U 19.7 18.5 3 3
521-543 caggttatcaacgaaacttctca AGAAGUUUCGUUGAUAACCUG GGUUAUCAACGAAACUUCUCA U 13.3 16.1 2 3
このようにsiDirectのweb版でテーブルとして表示される部分が、タブ区切りテキストで得られる。
siRNAを設計する際のオプションは、スクリプト内の %param で与えることができる。詳細は下の図およびコードを参照。
大量にクエリを投げる場合の例。サーバに負荷が掛からないよう、sleepコマンドをはさんでほしい。
% ls
input_sequences/ siDirect2crawl.pl* siDirect_result/
% cd input_sequences/
% ls
NM_000014.fa NM_000015.fa NM_000016.fa […]
% foreach n ( * )
foreach> ../siDirect2crawl.pl $n > ../siDirect_result/$n.siRNA
foreach> sleep 5
foreach> end
[…]
% cd ../siDirect_result/
% ls
NM_000014.fa.siRNA NM_000015.fa.siRNA NM_000016.fa.siRNA […]
- Comments (Close): 0
- Trackbacks: 0
RefExとの連携&データ出力機能を追加
- 2011-06-13 (月)
- DBCLS
RefExとの連携
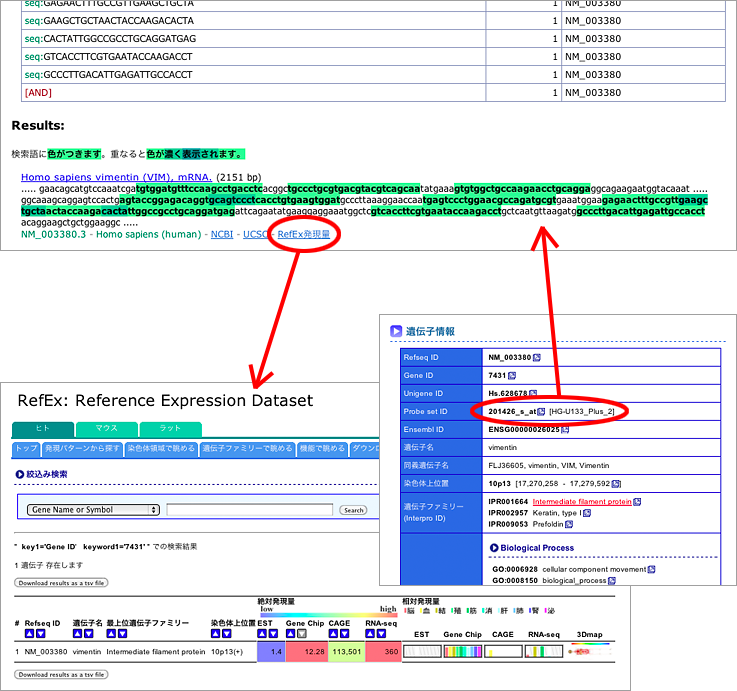
マイクロアレイのプローブIDから配列をつかって遺伝子を検索できるようにしたので、RefExからリンクを張っていただきました。RefExはDBCLSで提供している発現量のリファレンスデータセット。これで相互リンクが実現。同じ部屋の方の仕事とリンクできていい感じです。
データ出力機能
検索結果を外部のソフトで利用しやすいように、タブ区切りテキストで出力できるようにしました。
「タブ区切りテキスト。エクセルとかに直接コピペできます。エクセルはお嫌いですか?→ Numbersへ」
窓の中身を選択して、たとえばExcelに直接コピー&ペーストすれば
うまい具合にセルに入ります。 もちろんNumbersでもうまくいきますよ!!
- Comments (Close): 0
- Trackbacks: 0
アレイのプローブ配列検索を強化
- 2011-06-09 (木)
- DBCLS
マイクロアレイのプローブIDから塩基配列検索の機能を強化しました。
「便利そう」なサービスを実際に「使える」サービスにするためには、地味な作り込みが不可欠なんですよね・・・。先週紹介したプローブ配列検索も、ヒトとマウスで合計4つのプラットフォームだけでは頼りない。そこで今回は、AffymetrixとAgilentのマイクロアレイのうち、GEOにサンプル登録が多いプラットフォームと、まだサンプル登録は少ないけれども最新のプラットフォームを、とにかく全部収録することに。
収録したプラットフォーム一覧
| メーカー | 生物種 | マイクロアレイの種類 | GPL ID |
|---|---|---|---|
| Affymetrix | ヒト | Human Genome U219 Array | GPL13667 |
| Affymetrix | ヒト | Human Genome U133 Plus 2.0 Array | GPL570 |
| Affymetrix | マウス | Mouse Genome 430 2.0 Array | GPL1261 |
| Affymetrix | ラット | Rat Genome 230 2.0 Array | GPL1355 |
| Affymetrix | ラット | Rat Genome U34 Set (U34A/B/C) | GPL85,GPL86,GPL87 |
| Affymetrix | ニワトリ | Chicken Genome Array | GPL3213 |
| Affymetrix | ゼブラフィッシュ | Zebrafish Genome Array | GPL1319 |
| Affymetrix | ショウジョウバエ | Drosophila Genome 2.0 Array | GPL1322 |
| Affymetrix | ショウジョウバエ | Drosophila Genome Array | GPL72 |
| Affymetrix | 線虫 | C. elegans Genome Arra | GPL200 |
| Affymetrix | シロイヌナズナ | Arabidopsis ATH1 Genome Array | GPL198 |
| Affymetrix | 出芽酵母+分裂酵母 | Yeast Genome 2.0 Array | GPL2529 |
| Affymetrix | 出芽酵母 | Yeast Genome S98 Array | GPL90 |
| Agilent | ヒト | SurePrint G3 Human Exon 2x400K Microarray (028680) | |
| Agilent | ヒト | SurePrint G3 Human Exon 4x180K Microarray (028679) | |
| Agilent | ヒト | SurePrint G3 Human GE 8x60K Microarray (028004) | GPL13607 |
| Agilent | ヒト | Whole Human Genome Microarray 4x44K v2 (026652) | GPL10332,GPL13497 |
| Agilent | ヒト | Whole Human Genome Microarray 4x44K (014850) | GPL4133,GPL6480,GPL9822 |
| Agilent | マウス | SurePrint G3 Mouse Exon 2x400K Microarray (028727) | |
| Agilent | マウス | SurePrint G3 Mouse Exon 4x180K Microarray (030493) | |
| Agilent | マウス | SurePrint G3 Mouse GE 8x60K Microarray (028005) | GPL10787 |
| Agilent | マウス | Whole Mouse Genome Microarray 4x44K v2 (026655) | GPL10333,GPL11202 |
| Agilent | マウス | Whole Mouse Genome Microarray 4x44K (014868) | GPL4134,GPL7202 |
| Agilent | ラット | SurePrint G3 Rat Exon 2x400K (028728) | |
| Agilent | ラット | SurePrint G3 Rat Exon 4x180K (028744) | |
| Agilent | ラット | SurePrint G3 Rat GE 8x60K Microarray (028279) | |
| Agilent | ラット | Whole Rat Genome Microarray 4x44K v3 (028282) | |
| Agilent | ラット | Whole Rat Genome Microarray 4x44K (014879) | GPL4135,GPL7294 |
| Agilent | ニワトリ | G. gallus (Chicken) Oligo Microarray v2 (026441) | |
| Agilent | ニワトリ | Chicken Gene Expression Microarray (015068) | GPL8764 |
| Agilent | ゼブラフィッシュ | D. rerio (Zebrafish) Oligo Microarray V3 (026437) | |
| Agilent | ゼブラフィッシュ | D. rerio (Zebrafish) Oligo Microarray (013223) | GPL2878,GPL7244 |
| Agilent | ゼブラフィッシュ | Zebrafish (v2) Gene Expression Microarray (019161) | GPL6457,GPL7301 |
| Agilent | ゼブラフィッシュ | Zebrafish Gene Expression Microarray (015064) | GPL6563,GPL7302 |
| Agilent | ショウジョウバエ | D. melanogaster (FruitFly) Oligo Microarray - V2 (021791) | |
| Agilent | ショウジョウバエ | Drosophila Gene Expression Microarray (018972) | GPL6385,GPL7300 |
| Agilent | 線虫 | C. elegans Oligo Microarray (012795) | GPL2875,GPL7272 |
| Agilent | 線虫 | C. elegans (V2) Gene Expression Microarray (020186) | GPL10094,GPL11346 |
| Agilent | 線虫 | C. elegans Gene Expression Microarray (015061) | GPL7727,GPL8209 |
| Agilent | シロイヌナズナ | Arabidopsis 3 Oligo Microarray (012600) | GPL2871,GPL7270 |
| Agilent | シロイヌナズナ | Arabidopsis 2 Oligo Microarray (V2) (013324) | GPL2880,GPL7290 |
| Agilent | シロイヌナズナ | Arabidopsis 2 Oligo Microarray (011839) | GPL888,GPL7265 |
| Agilent | シロイヌナズナ | Arabidopsis (V4) Gene Expression Microarray (021169) | GPL9020,GPL12621 |
| Agilent | シロイヌナズナ | Arabidopsis (V3) Gene Expression Microarray (015059) | GPL6177,GPL7299 |
| Agilent | 出芽酵母 | Yeast microarray (011447) | GPL884,GPL7259 |
| Agilent | 出芽酵母 | Yeast Oligo Microarray (V2) (013384) | GPL2883,GPL7293 |
| Agilent | 出芽酵母 | Yeast (V2) Gene Expression Microarray (016322) | GPL9825,GPL10045,GPL11488,GPL13340 |
| Agilent | 出芽酵母 | Yeast (V1) Gene Expression Microarray (015072) | GPL7542,GPL9294 |
プローブ検索、既知の問題点
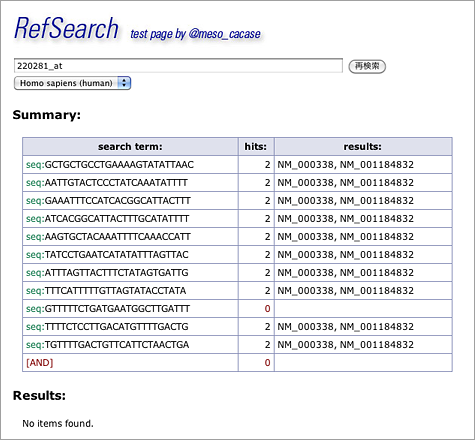
プローブを検索しても結果が何も出てこない場合があります。たとえばヒトの 220281_at というプローブ。
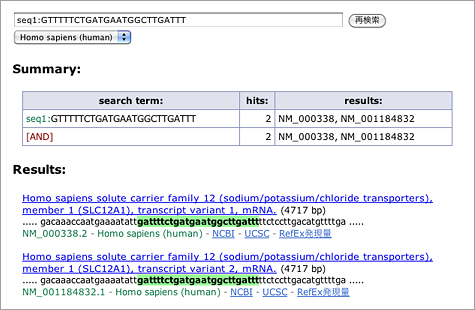
Summary欄の表をみると、11個あるプローブのうち10個は NM_000338, NM_001184832 にマッチしているものの、GTTTTTCTGATGAATGGCTTGATTT というプローブは何もヒットしていません。したがってANDを取ると何も出てこないことになります。ところがこのプローブ、1ミスマッチを許して seq1:GTTTTTCTGATGAATGGCTTGATTT で検索してやると(リンク)、ちゃんと NM_000338, NM_001184832 がヒットするのです。Affymetrix社がプローブ設計時に使った配列が、RefSeqの最新の配列と1塩基違っていたのですね。
こういう事例を想定して、本当は11個の塩基配列を検索するときに最初からミスマッチを許して seq1: とか seq2: のモードで検索すればよいのでしょうが、計算時間がかかるのでどうすべきか・・・。
- Comments (Close): 0
- Trackbacks: 0
マイクロアレイのプローブIDから塩基配列検索
- 2011-06-02 (木)
- DBCLS
地味ですが新しい機能を追加しました。マイクロアレイのプローブIDを入力すると、そのプローブの塩基配列をつかって遺伝子を検索してくれる機能です。
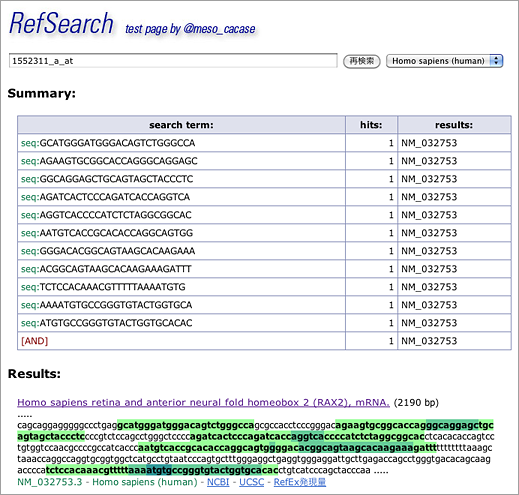
たとえば下の例は、Affymetrix社のヒトのアレイ(GeneChip Human Genome U133 Plus 2.0 Array)のプローブである、1552311_a_at で検索したものです(リンク)。Affymetrixのアレイは25-merのオリゴ11本で1つのtranscriptを認識するようになっています。この11本をまとてprobesetと呼び、probesetごとに上記のようなIDが振られています。そこで、
「1552311_a_at」
と入力すると、サーバ側でプローブID→塩基配列に展開し、
「GCATGGGATGGGACAGTCTGGGCCA」+
「AGAAGTGCGGCACCAGGGCAGGAGC」+
「GGCAGGAGCTGCAGTAGCTACCCTC」+
「AGATCACTCCCAGATCACCAGGTCA」+
「AGGTCACCCCATCTCTAGGCGGCAC」+
「AATGTCACCGCACACCAGGCAGTGG」+
「GGGACACGGCAGTAAGCACAAGAAA」+
「ACGGCAGTAAGCACAAGAAAGATTT」+
「TCTCCACAAACGTTTTTAAAATGTG」+
「AAAATGTGCCGGGTGTACTGGTGCA」+
「ATGTGCCGGGTGTACTGGTGCACAC」
で検索を実行するようにしてみました。結果はこちら。
Result欄を見ると、RAX2 (NM_032753) という遺伝子がヒットしていることがわかります。遺伝子のリンクをクリックすると、
のように、3’末端付近に11本のオリゴの標的サイトがあることがわかります。余談ですが、検索でヒットした文字列が重なった場合は緑色が濃くなるように改良しました。今までのように単色だと、どこがどのように重なっているのか全然わからなかったので。
ところで皆さんはマイクロアレイのデータを解析するときに、プローブIDと遺伝子をどのように対応させているでしょうか。おそらく多くの方は、メーカーが出している対応表や、アレイ解析ソフトの結果をそのまま利用されていると思います。しかしこれは意外と信用できないとmesoは考えています。
少し前の話なのですが、Affymetrixのアノテーションでは 226437_at → VIM とあるのに、データがおかしいのでよく調べてみたら、実際にはまったく別の遺伝子であるYIF1Bに当たっていた、みたいな事例が頻繁にありました。
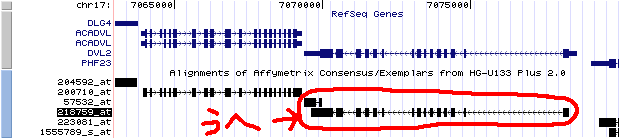
Affymetrixの表がちょっと軽くやばい感じだったので、Biomartを利用して対応表をつくることを試みたのですが、それもいまいち。例えば対応表のうえではACADVLに当たっているはずの3つのプローブが、よく調べてみると下の図のように3つのうち2つが隣の遺伝子(DVL2)に当たっていたり・・・。
上記の2つの事例に関しては、2011年6月現在は訂正されているものの、まだまだあやしい箇所が残っているのではないかと思います。だからこそ、ユーザ自身がプローブIDから塩基配列をつかって遺伝子(より正確にはtranscript)との対応を調べることは大事だと思うのです。現状ではそれを簡単にできる方法がなかったので、つくってみたというわけでした。今回は、
- GeneChip Human Genome U133 Plus 2.0 Array (Affymetrix)
- GeneChip Mouse Genome 430 2.0 Array (Affymetrix)
- Whole Human Genome Microarray 4x44K v2 (Agilent)
- Whole Mouse Genome Microarray 4x44K v2 (Agilent)
のデータを取り込んでおり、随時拡充していく予定です。
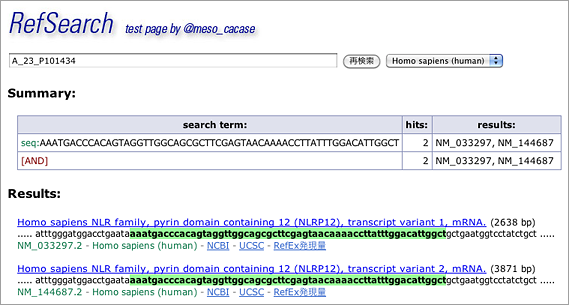
下記はAgilentのヒトのアレイ(Whole Human Genome Microarray 4x44K v2)から A_23_P101434 を検索した例(リンク)。Agilentのアレイは基本的には60-merのオリゴ1本で1つのtranscriptを認識するようにできている点がAffymetrixとの大きな違いです。mesoの経験的にはこちらのほうがバックが小さくなり、発現量が比較的少ない遺伝子まで綺麗にデータが出るようです。
- Comments (Close): 0
- Trackbacks: 0
生物種の追加とデータベース更新
- 2011-05-31 (火)
- DBCLS
前回のpostでは、mesoが構築している「遺伝子をGoogleのように(?)検索できるサイト」を紹介した。
http://bit.ly/RNAbrowser→ http://GGRNA.dbcls.jp/ (8/18更新:URLを変更)
まずはヒト遺伝子バージョンを作って公開したわけだが、それだけではヒトをやっている人にしか使ってもらえないので生物種を増やすことにした。とは言ってもマシンの容量の制約があるので、取り入れる生物種を絞らないといけない。
生物種の選定
今回はmesoの独断と偏見に基づき、モデル生物として普及しており、かつゲノムや個々の遺伝子に関する情報が豊富な生物種を10種くらい選ぶことにした。@synobuさんが教えてくれたGene Ontologyコンソーシアムのページも参考にした。
- GO: Reference Genome Annotation Project – http://www.geneontology.org/GO.refgenome.shtml
このページの中ほどにあるモデル生物のリストが使えそう。
- Arabidopsis thaliana – シロイヌナズナ
- Caenorhabditis elegans – 線虫
- Danio rerio – ゼブラフィッシュ
- Dictyostelium discoideum – タマホコリカビ(今回は見送り)
- Drosophila melanogaster – ショウジョウバエ
- Escherichia coli – 大腸菌(今回は見送り)
- Gallus gallus – ニワトリ
- Homo sapiens – ヒト
- Mus musculus – マウス
- Rattus norvegicus – ラット
- Saccharomyces cerevisiae – 出芽酵母
- Schizosaccharomyces pombe – 分裂酵母
このうち大腸菌、タマホコリカビ、それから別にリクエストをいただいた枯草菌、シアノバクテリアは、データの形式の問題で今回は見送り、別に対応を考えることにした。またtwitterでご連絡いただいたイネをはじめNBRPな生物種は次回以降の更新でぜひ取り入れたい。
データの取得
ちょうど5月に入ってRefSeq release 47がリリースされたので、ついでにヒトの配列も更新する。RefSeqは2ヶ月に1回の頻度で新しいバージョンがリリースされるので、mesoの検索サイトもそれにあわせて更新できるよう、データベースの追加・更新を簡単にできるような仕組みをつくる必要がある。
RefSeqの最新版は下記からダウンロードできる。
この中にある全生物種の転写産物を収録したGBFF(GenBank Flat File)形式のファイル、completeXXX.rna.gbff.gz(release 47ではXXXは1〜418)をダウンロードした。容量はgz圧縮で3.4GB、展開すると17.2GBある。各transcriptのSOURCE欄を見て特定の生物種だけを抜き出すスクリプト(grep_gbff.pl)を使い、前述の10種を抜き出した。
| 生物種 | GenBank形式(GBFF) ファイルのSOURCE欄 | Transcript数 | GenBank形式(GBFF) ファイルの容量(MB) |
|---|---|---|---|
| ヒト | Homo sapiens (human) | 46,727 | 846.6 |
| マウス | Mus musculus (house mouse) | 35,892 | 585.4 |
| ラット | Rattus norvegicus (Norway rat) | 30,862 | 225.0 |
| ニワトリ | Gallus gallus (chicken) | 5,418 | 38.8 |
| ゼブラフィッシュ | Danio rerio (zebrafish) | 28,261 | 190.5 |
| ショウジョウバエ | Drosophila melanogaster (fruit fly) | 22,929 | 352.1 |
| 線虫 | Caenorhabditis elegans | 24,377 | 165.0 |
| シロイヌナズナ | Arabidopsis thaliana (thale cress) | 34,525 | 197.0 |
| 出芽酵母 | Saccharomyces cerevisiae S288c | 5,863 | 37.8 |
| 分裂酵母 | Schizosaccharomyces pombe 972h- | 5,010 | 39.6 |
| 合計 | 239,864 | 2677.7 |
検索サイト更新
以上のデータを取り入れて検索サイトをアップデートした。当初はあくまで「窓ひとつ」で検索できることにこだわっており、spe:human のように生物種もタグで指定することを考えていたが、それはやめてプルダウンで選ぶ方針にした。ユーザの立場で考えてみると、検索の時点で生物種が決まっている場合が多いだろうということと、普段ショウジョウバエなど特定のモデル生物を扱っている研究者は、その種を中心に検索することが多いという事情を考慮した。
どうせなら入口を分けてもよいかもしれない。下記のURLからアクセスすると、最初からそれぞれの種がプルダウンで選ばれた状態になる。毎回プルダウンを選択する必要がないうえに、ブックマークもしやすいだろう。
- ヒト → http://GGRNA.dbcls.jp/hs/
hsを省略して http://GGRNA.dbcls.jp/ だけでもOK - マウス → http://GGRNA.dbcls.jp/mm/
- ラット → http://GGRNA.dbcls.jp/rn/
- ニワトリ → http://GGRNA.dbcls.jp/gg/
- ゼブラ → http://GGRNA.dbcls.jp/dr/
- ハエ → http://GGRNA.dbcls.jp/dm/
- 線虫 → http://GGRNA.dbcls.jp/ce/
- シロイヌナズナ → http://GGRNA.dbcls.jp/at/
- 出芽酵母 → http://GGRNA.dbcls.jp/sc/
- 分裂酵母 → http://GGRNA.dbcls.jp/sp/
(6/1更新)URLを少し変更して /~meso/ なしでアクセスできるようにしました。過去にアナウンスしたURLも当分は使えますが、上記のURLをご利用ください。
(8/18更新:URLを全面的に変更しました。過去にアナウンスしたURLから転送されます。
ちなみにこれはウェブサーバ側でURLの /mm/ の部分を index.cgi?spe=mm などと書き換えるRewriteRuleを書いて実現している。mod_rewrite便利。
さて、こうしているうちに新たなバグが見つかったので修正しないと。
- Comments (Close): 0
- Trackbacks: 0
遺伝子をGoogleのように検索できるサイト
- 2011-05-18 (水)
- DBCLS
遺伝子をGoogleのような感覚でサクサク検索できたら便利だろうと考え、サイトをつくってみた。mesoが構築している「RNA統合データベース」を構成する仕組みのひとつとなる予定だ。
http://bit.ly/RNAbrowser→ http://GGRNA.dbcls.jp/ (8/18更新:URLを変更)
NCBI RefSeq release 46 (Mar, 2011)に登録されているヒトのtranscriptを対象にフリーワードで検索できる。とくに、塩基配列まで含めて検索できるようにしたところがこのサイトの売りで、オプションにより1〜2塩基のミスマッチを許した検索も可能だ。
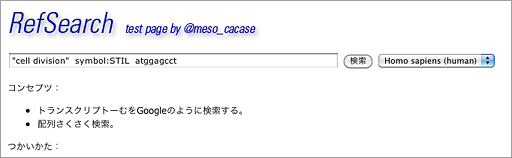
使いかたは、検索ウィンドウに単語を打ち込むだけ。あえて入り口はシンプルにした。細かい条件をつけたいときのために、aa:KLQEEM(アミノ酸配列から検索)のようなタグを何種類か用意した。ただしタグを明示的につけなくても多くの場合は望みの結果が得られるはずだ。
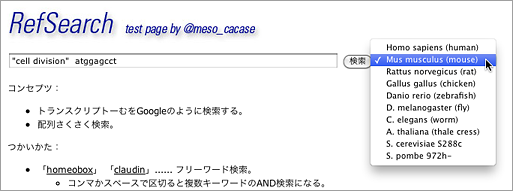
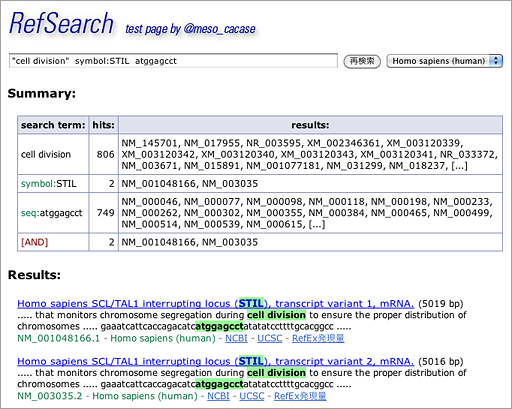
複数のキーワードを入れるとAND検索になり、たとえばこんな結果が表示される。
上の例では、”cell division”というキーワードで806件、symbol:STIL(symbolから検索)で2件、atggagcctという塩基配列を持つものが749件。そして、3つの検索語をすべて含むものが2件([AND]欄を参照)見つかった。Results:欄にその2件の遺伝子の概要が表示されている。
(5/26追記)「2件の遺伝子」と書いてしまったが、この2件はSTILという1つの遺伝子に由来する2種類のtranscript variantである。
このように複数のキーワードを入れた場合は、いわゆる絞り込み検索をするのではなく、3つの独立した検索をおこなってその結果の共通部分を求める仕組みにした。これは、各々の検索語ごとのヒット件数がわかるほうが便利だろうと思ったからだ。たとえば、ユーザの意図する検索結果が出てこなかった場合、検索語を変えたうえで再検索することになるわけだが、どの検索語を変えればうまく絞り込めるのかを考える上で参考になるだろう。
キーワードごとに独立した検索をおこなっているため時間はかかるが、それでも上記の結果は約1秒で返ってくる。
さて、Results:欄だが、NCBIのnucleotide、UCSCゲノムブラウザ、RefExにリンクしている。RefExはDBCLSで提供している発現量のリファレンスデータセットだ。検索でヒットした遺伝子がどのくらい発現しているのかをワンクリックで概観できる。
- RefEx — http://togoexp.dbcls.jp/aboutrefex
DBCLSが提供している遺伝子の発現量データベース。
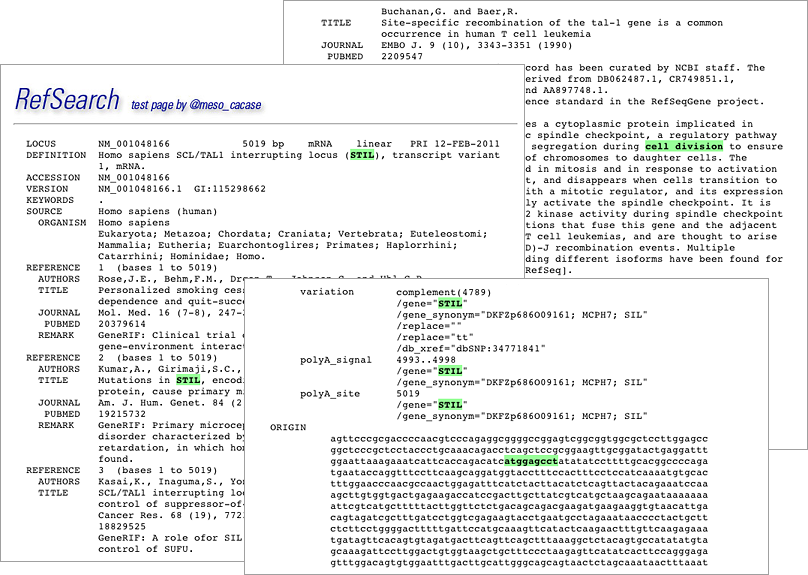
さらに、Results:欄の各遺伝子のリンクをクリックすると、おなじみのGenBankフォーマットで詳細が表示される。入力したキーワードがハイライトされ、見つけやすくなっている。
開発途上のため不安定なところがあるかもしれない。お手数でなければ不具合のご指摘やコメントなど頂ければたいへんありがたい。
将来は、転写産物=RNAをベースに知識(塩基配列、文献、プローブ、siRNA、等々)を統合したデータベースをつくり、さまざまな切り口で遺伝子を「さがす」または「ながめる」ことができるようなサイトの公開を目指している。何をどんなふうに統合して、どんな使い勝手になるのかは別に紹介していきたいと思う。今回はその準備という位置づけだ。
なお今回のサイトは、2006年頃に私がPreferred Infrastructureの西川徹さんらと一緒にコンセプトや仕様を考え、西川さんらが実装したGenome Sedueというサイトをもとにして、RNA統合データベースに必要な仕組みを盛り込んで発展させている。
- Preferred Infrastructure — http://preferred.jp/
検索エンジン関連を中心に非常に高い技術をもつ会社。 - Genome Sedue — http://labs.preferred.jp/genome/
現在は使えなくなっている。
- Comments (Close): 0
- Trackbacks: 0
ウェブサービスの立ち上げとXSS対策
- 2011-04-27 (水)
- DBCLS
mesoがDBCLSに着任してまずやろうとしていることは、転写産物=RNAベースで、塩基配列、文献、プローブ、siRNA、等々を整理した統合データベースをつくることだ。従来のデータベースの多くは何でもゲノムにマッピングするという発想だったが、biologistはどちらかというと遺伝子や転写産物を単位にものごとを考えているはずなので、転写産物に知識をマッピングするほうが自然だろう。しかも、それをフリーワードや塩基配列からサクサク検索できるサイトをつくれば有用に違いない。
プロトタイプができたらここで紹介する予定だが、今日はグループのメンバーに作成途中のwebを回覧したところ、即座にXSSを仕掛けられて脆弱性が露見するという情けないことがおきたので対策を施すことにした。
XSSとは
XSS(クロスサイトスクリプティング)とは何か。例を挙げると、ウェブの入力フォームに、お名前:「<script>alert(“うへへ”)</script>」のようないじわるな文字列を入力すると、返ってきたHTMLに含まれる「ようこそ、○○さん」のところが「ようこそ、<script>alert(“うへへ”)</script> さん」になりJavaScriptとして解釈されてしまう、こういった脆弱性を突く行為だ。悪用されれば深刻な被害が出かねないので対策が必須である。詳しくは、今回参考にした「@IT: クロスサイトスクリプティング対策の基本」などを参照してほしい。
mesoの作成しているページには早速 @iNut たんがあやしいクエリをsubmitしたところ、下記のようにJavaScriptのalert()が実行されて奇妙なポップアップが表示された。
駄目だこれは(=_=。ちなみに@iNut様のブログ、Chateau Togo によると、うちのグループのスタンダードは、
僕がこの牧場で教わったことは3つありますー
一つ、書いたコードは恥を承知で晒せ。
一つ、コマンドの使い方が分からなければmanを読め。
一つ、フォームを見たらとりあえずXSSを打ち込め。
ということらしい。meso涙目。
XSS対策
XSS対策をする場所は基本的に2箇所。入力データのチェックと、HTML出力前のチェックだ。下記の特殊文字をHTMLの実態参照に置き換えるか、消去してしまえば、タグとして解釈されなくてすむ。これをサニタイズ(無害化)する、と言うらしい。
HTMLのタグとして解釈される可能性がある文字:
- 「<」→ <
- 「>」→ >
- 「&」→ &
タグ中で<a href=”hoge”>のような属性値の開始・終了に使われる文字:
- 「’」→ '
- 「”」→ "
ただ、上記のような置き換えを考える前に、本当にそんな文字を受け付ける必要があるのか? を検討するべきだろう。たとえば郵便番号は半角数字3桁+4桁と決まっているので、それ以外の文字列は入力の時点でチェックしてはねるのが先決だ。入力のチェックを厳しくすることは、XSS以外の脆弱性(オーバーフロー等)の対策にもなる。
なお、ここに書いたことはあくまで基本なのでこれだけでは対策として不十分だが、何もしないのと比べると大幅にリスクを減らすことができるだろう。詳しくは「XSS 対策」でググってさらに調べるべし。
実験用のコードとサービス用のコードは違う
とりあえず上記+αの対策で@iNutさんの意地悪クエリには対処できた。しかしこんどは @chalkless さんから、あるクエリを投げるとDBのほぼ全件が返ってくるとか、長いクエリを投げるとオーバーフローするといった指摘をいただいた。meso涙がとまらない。
あたりまえのことだが、実験のために書くコードと、サービス提供のためのコードは違うのである。すべての条件分岐と、すべてのパラメータをチェックし、どんな入力を与えても(あるいは与えなくても)エラーで落ちないように書かなければいけない。たとえばperlのサブルーチンなどでよく出てくる、
my $query = $_[0] ;
というところは、
my $query = $_[0] || ‘デフォルト値’ ;
# 注)$_[0]がundef, 空白, ゼロだと「偽」が返るので || (or) の右項が評価され、
# $queryに ‘デフォルト値’ が入る。
などと書いておかないとundefとか空白だった場合にしぼうするだろうし、状況によって成功しないかもしれない文を実行するときには、
eval(実行したい文) or print_error(‘○○できませぬ!’) ;
# 注)evalの中身の文がコケてもプログラム自体は止まらずevalがゼロを返す。
のように「保険をかける」書き方が必要になってくる。また直接的な対策ではないが、デバッグしやすいコードを書く(例えばサブルーチン化を徹底する等)ことも、セキュリティ対策に寄与するだろう。(このへんの考え方は、メガバンクの勘定系システムをやっていたココ殿から昔レクチュアしていただいたことがある。銀行システムは超ガチガチで大変だったらしいけど・・・)
セキュリティ対策がある程度できたら、作成中のサイトをこの場で紹介したい。
- Comments (Close): 0
- Trackbacks: 0
Mac OS Xのコマンドラインでファイルを完璧にコピーする方法
- 2011-04-21 (木)
- DBCLS
Mac OS XのファイルはリソースフォークとかExtended Attribute、ACLといった情報を含んでおり、単なるcpとかscpではこのような情報が完璧に保持されるわけではない。
以前にMac OS X 10.5(Leopard)のコマンドラインで「完璧な」ファイルのコピーをおこなう方法を検討したのでその方法を書き留めておく。なお、Mac OS X 10.6(Snow Leopard)に移行してもそのまま利用可能だ。
基本的にはrsyncを使っている。rsyncのメリットは何と言っても差分コピーができること。たとえばホームディレクトリの定期的なバックアップに利用すれば、変更したファイルだけをコピーしてくれるので無駄がない。ファイルに変更がなくても、たとえばパーミッション等を変えるとちゃんとその情報がコピーされるのも隙がない。
もうひとつのメリットは、sshを利用して別のマシンに「完璧な」コピーを送れること。複数のMacをまったく同じ環境にセットアップしたり、リモートからバックアップを取るのに役立つ。ちなみにmesoは職場と自宅のMacを毎日この方法で同期していた。差分だけを転送するので数分程度で完了する(ただし巨大なファイルを転送するときは別)。
ここで注意が必要なのは、Leopardに標準で入っているrsyncは、どういうわけか毎回全てのファイルのリソースフォークをコピーして効率が悪いうえ、ハードリンクなどが保持されない。そこでrsyncにいくつかのパッチをあてソースからコンパイルすることにした。
参考
- Mac OS X and HFS+ enhancements for rsync 3(rsyncをMac OS X用に拡張する詳細な解説)
- rsyncの公式ページ
- Compiling Rsync 3 as a Universal Binary for Leopard
rsyncのコンパイル
curl -sO http://rsync.samba.org/ftp/rsync/src/rsync-3.0.4.tar.gz curl -sO http://rsync.samba.org/ftp/rsync/src/rsync-patches-3.0.4.tar.gz tar xzf rsync-3.0.4.tar.gz tar xzf rsync-patches-3.0.4.tar.gz cd rsync-3.0.4 # Apply patches relevant to preserving Mac OS X metadata patch -p1 < patches/fileflags.diff patch -p1 < patches/crtimes.diff ./configure make
成功すればrsyncというバイナリができているはずだ。なお、ACLは10.4(Tiger)以降なので、Mac OS 10.3(Panther)上ではACL関係でエラーが出てコンパイルできない。
mesoは職場でIntel Mac、自宅でPowerPC Macを使っているので、両方の環境で実行できるようにユニバーサルバイナリ化した。
# Intel Macでmakeしたもの → rsync-3.0.4-i386/rsync # PowerPC Macでmakeしたもの → rsync-3.0.4-PPC/rsync # として、lipoでユニバーサルバイナリ化する。 lipo -create rsync-3.0.4-i386/rsync rsync-3.0.4-ppc/rsync -output rsync # OS標準のrsyncと容易に区別できるよう、名前を変えておく。 cp -pi rsync ~/bin/rsync-304
Backup Bouncerによる評価
上記でmakeしたrsyncで「完璧な」コピーができるのかを、Backup Bouncerで検証。Backup Bouncerは、Mac OS Xのファイルが保持している各種情報が、コピー後にもちゃんと継承されているかどうかをテストするためのツール。ここで評価したのは、
- Mac OS X 10.5 (Leopard)標準のrsync
- rsyncx
- rsync_hfs
- 今回makeしたrsync-304
結果のみ示す。すべてのテスト項目をパスしたら、とりあえず完璧と呼ぶことにする。
| テスト項目 | Leopard rsync | rsyncx | rsync_hfs | rsync-304 | ←304で使用 するオプション |
|---|---|---|---|---|---|
| basic-permissions (Critical) | ok | ok | ok | ok | -a |
| timestamps (Critical) | ok | ok | ok | ok | -a |
| symlinks (Critical) | ok | ok | ok | ok | -a |
| symlink-ownership | ok | FAIL | FAIL | ok | -a |
| hardlinks (Important) | FAIL | FAIL | FAIL | ok | -aH |
| resource-forks, on files (Critical) | ok | ok | ok | ok | -aX |
| resource-forks, on hardlinked files (Important) | FAIL | FAIL | FAIL | ok | -aHX |
| finder-flags (Critical) | ok | FAIL | FAIL | ok | -aX |
| finder-locks | FAIL | ok | FAIL | ok | –fileflags |
| creation-date | FAIL | ok | FAIL | ok | -aN |
| bsd-flags | ok | ok | ok | ok | -a |
| extended-attrs, on files (Important) | ok | FAIL | FAIL | ok | -aX |
| extended-attrs, on directories (Important) | ok | FAIL | FAIL | ok | -aX |
| extended-attrs, on symlinks | FAIL | FAIL | FAIL | ok | -aX |
| access-control-lists, on files (Important) | ok | FAIL | FAIL | ok | -aA |
| access-control-lists, on dirs (Important) | ok | FAIL | FAIL | ok | -aA |
| fifo | ok | ok | ok | ok | -a |
| devices | ok | FAIL | ok | ok | -a |
| combo-tests, xattrs + rsrc forks | ok | FAIL | FAIL | ok | -aX |
| combo-tests, lots of metadata | ok | FAIL | FAIL | ok | -aAX |
rsync-304のつかいかた
今回makeしたrsync-304ですべてのテストをパスすることがわかったので、これを使っていくことにした。与えるオプションは次の通り。
rsync-304 -avNHAX --fileflags --force-change --delete --stats
別のマシンにコピーするときは、そのマシンにも今回makeしたrsync-304を入れておく必要がある。そして、相手側のrsyncの位置をオプションで与える。たとえば、
--rsync-path=/Users/meso/bin/rsync-304
以上をまとめて、mesoは下記のようにscpxというエイリアスを設定し、コピーはすべてこれに任せている。
alias scpx='/Users/meso/bin/rsync-304 \ --rsync-path=/Users/meso/bin/rsync-304 \ -avNHAX --fileflags --force-change --delete --stats'
つかいかた:
# デスクトップにコピー scpx hogehoge ~/Desktop/ # 別のマシンのデスクトップにコピー scpx hogehoge meso@133.11.XX.XX:Desktop/ # 別のマシンにあるディレクトリをlocalにコピー scpx meso@133.11.x.x:Desktop/hogehoge ./ # sshのポートを変更 scpx -e "ssh -p 12345" hogehoge meso@133.11.x.x:Desktop/ # ふつうにscpすると古いファイルは上書きされたり消されたり # するが、obsolete以下に移動して取っておくにはこうする。 # 差分バックアップで過去のファイルを残したいときに便利。 scpx --backup --backup-dir=obsolete meso backup/
- Comments (Close): 0
- Trackbacks: 0
WordPressのインストールとblog開設
- 2011-04-18 (月)
- DBCLS
@meso_cacase は4/1付でライフサイエンス統合データベースセンター(DBCLS)に着任しました。wetな実験からは離れますが、データベースやソフトウェア開発の面から生命科学に大きく貢献できるような仕事をしていきたいと思っています。引き続きよろしくお願いいたします。
さて、まずは @twittoru さんの記事「麻婆豆腐新劇場版 YOU ARE (NOT) ANNINDOFU.」を参考にg86にwordpressをインストールしてみる。余談だが、こういうときに「書き留め、共有する」が積極的に実践されている環境は素晴らしいと思う。
curl -sO http://ja.wordpress.org/latest-ja.tar.gz
展開して ~/Sites/meme/ に設置。各ファイルのパーミションに注意。mesoはデフォルトで umask 077 しているのでこのまま解凍するとウェブ経由で読めない。umask 000 した後に tar xzf すればOK。続いて、@iNut さんにmysqlのユーザを作ってもらう。
% /opt/local/bin/mysql5 -umeso -p
Enter password: (パスワードを入力)
mysql> CREATE DATABASE meso_wp ;
mysql> quit ;
meme/ 内の wp-config-sample.php を wp-config.php にコピーして下記のように変更。
// ** MySQL 設定 – こちらの情報はホスティング先から入手してください。 ** //
/** WordPress のためのデータベース名 */
define(‘DB_NAME’, ‘meso_wp’);/** MySQL データベースのユーザー名 */
define(‘DB_USER’, ‘meso’);/** MySQL データベースのパスワード */
define(‘DB_PASSWORD’, ‘パスワードを記載’);/** MySQL のホスト名 */
define(‘DB_HOST’, ‘localhost’);
あとはブラウザで / にアクセス。ガイダンスにしたがい若干の設定をすると完了。外観を簡単に変更できるらしい。「wordpress テーマ」でググるといろいろ出てくるが、ナイスなテーマを探していると1日つぶれてしまうので要注意。今回は wp.Vicuna というテーマを使ってみることにする。
さて、準備が整った。いままでDokuWikiを使ったことがあるので何となく勝手はわかる。今後はここを「実験ノート」として使い、「書き留め、共有する」ことを実践していこう。
(追記)
http://g86.dbcls.jp/~meso/ をwordpressのページ / にリダイレクトしてみる。.htaccessに
Redirect permanent /~meso/index.html /~meso/meme/
と書けばよいらしい。
- Comments (Close): 1
- Trackbacks: 0
- Search
- Feeds
- Meta